Kapitel 15 Lineares Modell - polynomiale Effekte und Interaktionen
15.1 Theorie
Bisher haben wir uns nur mit sogenannten Haupteffekten beschäftigt. Dies sind Effekte der Form \(\hat{\beta} \cdot x\).
In der Praxis sollte man immer auch prüfen, ob sich die Daten nicht durch eine komplexere Funktion besser (AIC/BIC) beschreiben lassen. So ist Wohlbefinden nicht linear mit Einkommen verknüpft, Pflanzenwachstum nimmt nicht linear mit Temperatur zu und die Anzahl von Gebäuden in einer Stadt ist nicht unbedingt eine lineare Funktion der Einwohneranzahl. Die einfachsten Optionen, dass zu tun, ist es Polynome höherer Ordnung71 oder Interaktionen zwischen den Prädiktoren ins Modell mit einzubeziehen.
Bei ökologischen Beziehungn finden sich z.B. oft Zusammenhänge, bei denen es eine optimale ökologische Nische gibt, z.B. bei den Dengue, Zika und Chikunguya übertragenden Tigermücken Aedes Aegypty und Aedes Albopictus, die hinsichtlich Temperatur einen Optimalbereich haben. Wird es kälter oder wärmer, ist die Mücke weniger aktiv und hat auch geringe Wahrscheinlichkeit für das Auftreten. Ein solcher Zusammenhang lässt sich durch einen quadratischen Term mit negativem Vorzeichen abbilden (s. linker Plot in Abbildung 15.1).
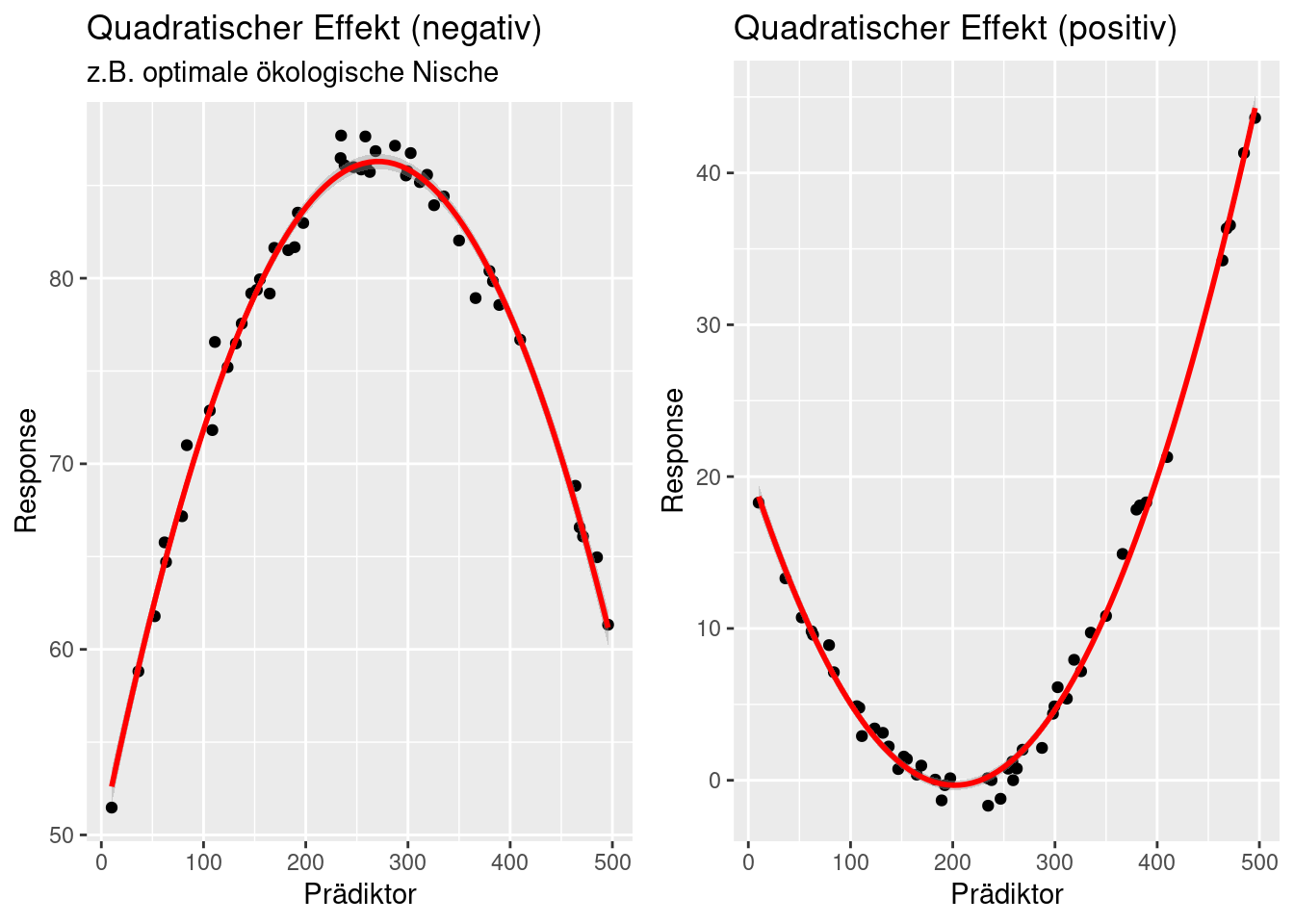
Figure 15.1: Quadratische Effekte.
Interaktionen beschreiben Situationen, bei denen der Effekt eines Prädiktors vom Wert eines anderen Prädiktors abhängt. Ein einfaches Beispiel ist Pflanzenwachstum. Pflanzen wachsen besser, wenn die Temperatur zunimmt 72, Pflanzen wachsen auch besser, wenn mehr Wasser verfügbar ist73. Die Effekte sind jedoch nicht additiv. Wenn wir 30°C haben und kein Wasser ist der Effekt ein anderer als bei 30°C und ausreichend Wasser.
Während man bei rein additiven Haupteffekten den Zusammenhang zwischen 2 Prädiktoren und dem Response nur mit einer ebenen Fläche, die man im Raum rotiert darstellen kann, erlauben Interaktionen es, die Regressionsfläche zu verbiegen.
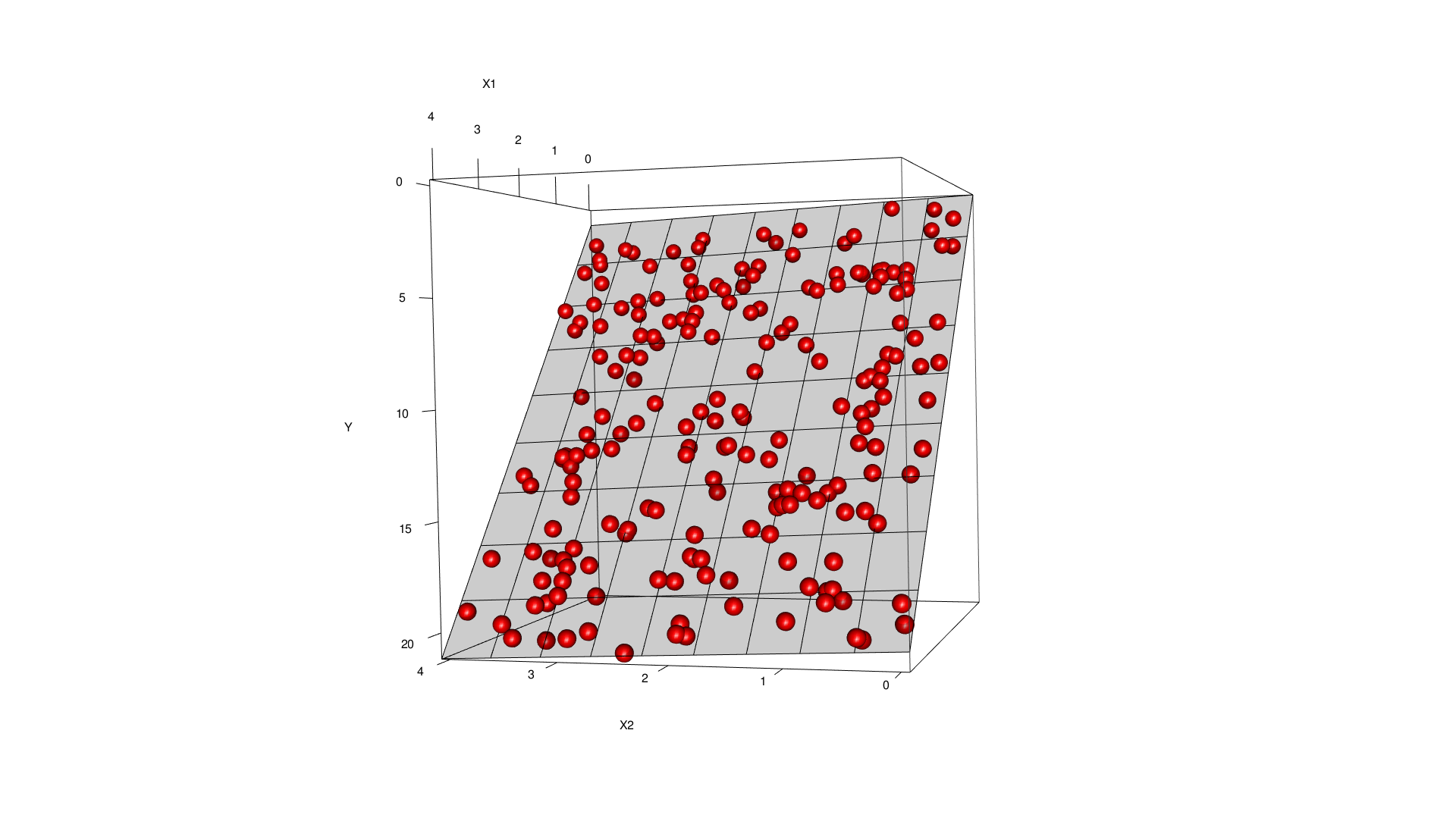
Figure 15.2: Regressiosnfläche für 2 Haupteffekte. Die Regressionsfläche ist eben, die Steigung kannn über die beiden Regressionskoeffizienten verändert werden..
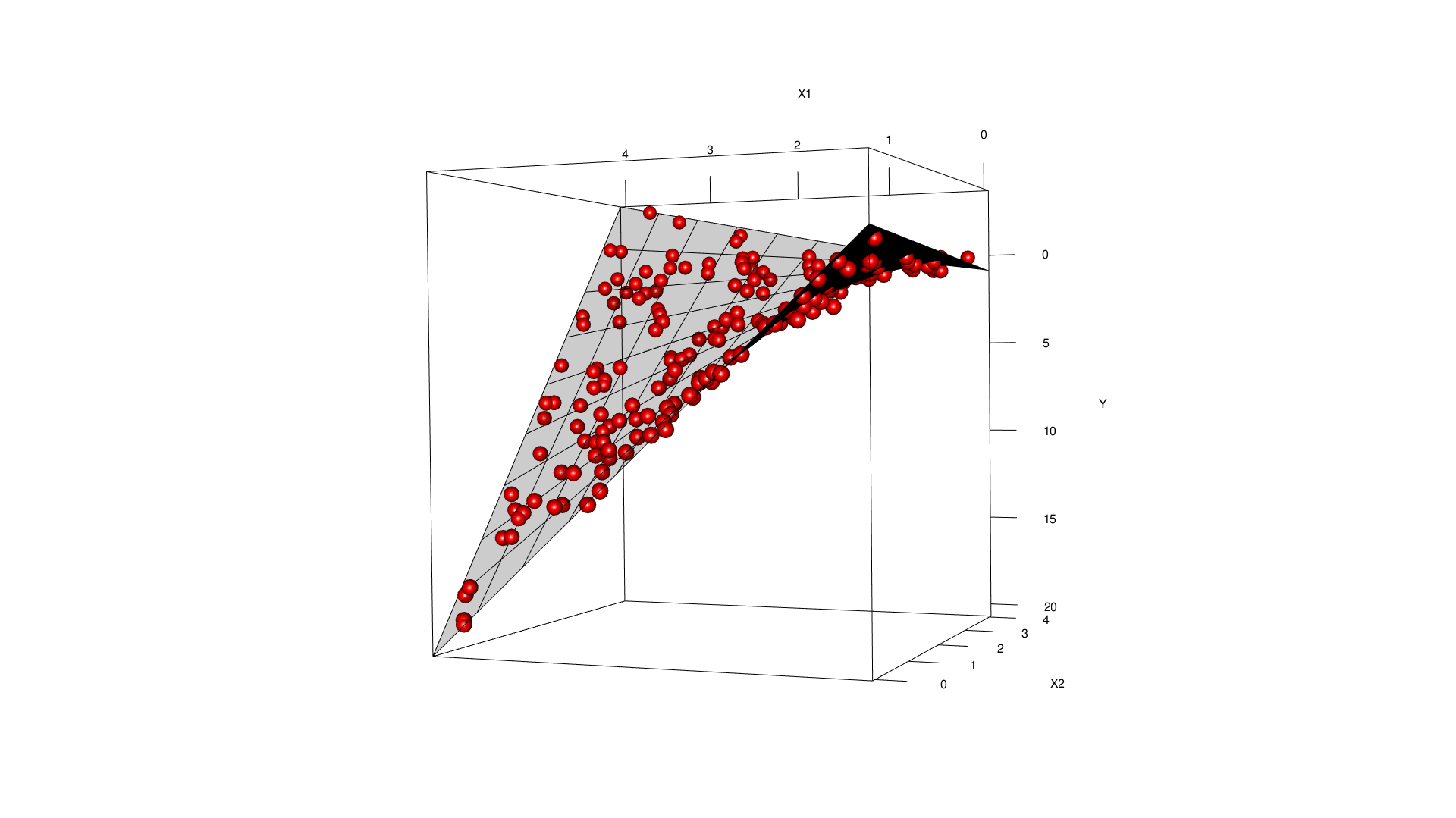
Figure 15.3: Interaktion zwischen den beiden Prädiktoren im Modell. Die Regressionsfläche ist gebogen.
15.2 Anwendungsbeispiel Temperaturdaten
Daten einlesen
15.2.1 Russland
Selektion von Stationen in Russland ohne NAs:
## [1] 50 36Standardisieren von Länge und Breite:
temp.ru$LAT.cent <- temp.ru$LAT - mean(temp.ru$LAT)
temp.ru$LON.cent <- temp.ru$LON - mean(temp.ru$LON)15.2.1.1 Polynomiale Effekt
Was ist mit quadratischen Effekten? Vielleicht wirken Längen- und Breitengrad ja auch quadratisch auf die Temperatur ein. Da der ^ Operator wir der + und der - und der * Operator überladen sind müssen wir den quadratischen Effekt entweder über I() oder über poly definieren. poly erzeugt dabei orthogonale Polygone, was i.d.R. von Vorteil ist.
Der Weg über I():
##
## Call:
## lm(formula = YEAR ~ ELEV + I(ELEV^2) + LAT.cent + LON.cent, data = temp.ru)
##
## Residuals:
## Min 1Q Median 3Q Max
## -2.60096 -0.78573 0.06268 0.85677 2.36774
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -2.439e-01 3.184e-01 -0.766 0.447807
## ELEV -5.388e-03 1.387e-03 -3.884 0.000334 ***
## I(ELEV^2) 3.719e-07 7.858e-07 0.473 0.638318
## LAT.cent -7.845e-01 3.516e-02 -22.309 < 2e-16 ***
## LON.cent -1.083e-01 1.154e-02 -9.384 3.66e-12 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.252 on 45 degrees of freedom
## Multiple R-squared: 0.9337, Adjusted R-squared: 0.9278
## F-statistic: 158.4 on 4 and 45 DF, p-value: < 2.2e-16Und nun über orthogonale Polynome mittels poly():
##
## Call:
## lm(formula = YEAR ~ poly(ELEV, 2) + LAT.cent + LON.cent, data = temp.ru)
##
## Residuals:
## Min 1Q Median 3Q Max
## -2.60096 -0.78573 0.06268 0.85677 2.36774
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -1.98438 0.17701 -11.211 1.29e-14 ***
## poly(ELEV, 2)1 -14.82349 1.52082 -9.747 1.15e-12 ***
## poly(ELEV, 2)2 0.65676 1.38774 0.473 0.638
## LAT.cent -0.78446 0.03516 -22.309 < 2e-16 ***
## LON.cent -0.10827 0.01154 -9.384 3.66e-12 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.252 on 45 degrees of freedom
## Multiple R-squared: 0.9337, Adjusted R-squared: 0.9278
## F-statistic: 158.4 on 4 and 45 DF, p-value: < 2.2e-16Wie wir sehen, erhalten wir deutlich andere Koeffizientenschätzer bei den beiden Ansätzen.
Wir wollen hier zunächst einfach bei den nicht orthogonalen Polygonen bleiben. Um festzustellen, ob der quadratische Term das Modell signifikant verbessert verwenden wir die Hilfsfunktion drop1, der jeden möglichen Term entfernt und jeweils den AIC (oder den BIC, wenn k entsprechend gesetzt ist) berechnet.
## Single term deletions
##
## Model:
## YEAR ~ ELEV + I(ELEV^2) + LAT.cent + LON.cent
## Df Sum of Sq RSS AIC
## <none> 70.50 27.177
## ELEV 1 23.64 94.13 39.635
## I(ELEV^2) 1 0.35 70.85 25.425
## LAT.cent 1 779.67 850.17 149.671
## LON.cent 1 137.96 208.46 79.386Wir sehen, dass der AIC um gut 2 Einheiten sinkt, wenn wir den quadratischen Term wieder entfernen. Ein deltaAIC von weniger als 2 (d.h. die Differenz zwischen 2 AIC Werten ist kleiner als 2) kann man als statistisch gleichwertige Modellgüten ansehen. Beide Modelle sind also in etwa gleich gut. Ich würde trotzdem das einfachere Modell bevorzugen.
Wichtig ist, das drop1 bei den quadratischen Effekten nicht automatisch das Marginalitätsprinzip respektiert. Wir müssen selbst dafür sorgen, dass es eingehalten wird, in dem wir keinen Haupteffekt aus dem Modell werfen, der in einem Term höherer Ordnung (oder einer Interaktion) enthalten ist.
Nun noch einaml mit dem BIC:
## Single term deletions
##
## Model:
## YEAR ~ ELEV + I(ELEV^2) + LAT.cent + LON.cent
## Df Sum of Sq RSS AIC
## <none> 70.50 36.737
## ELEV 1 23.64 94.13 47.283
## I(ELEV^2) 1 0.35 70.85 33.073
## LAT.cent 1 779.67 850.17 157.319
## LON.cent 1 137.96 208.46 87.034Hier ist das Ergebnis eindeutig: der quadratische Term, trägt nicht signifikant zur Modellgüte bei.
Nun noch einmal für die orthogonalen Polygone - die Interpretation ist hier schwieriger, da die Gleichung nicht mehr einfach \(a + bx + cx^2\) ist, sondern zwei neue orthogonal zueinander stehende Variablen x’ und x’’ erzeugt werden. Dafür sind beide Koeffizienten nicht miteinander korreliert (orthogonal).
## Single term deletions
##
## Model:
## YEAR ~ poly(ELEV, 2) + LAT.cent + LON.cent
## Df Sum of Sq RSS AIC
## <none> 70.50 27.177
## poly(ELEV, 2) 2 154.83 225.33 81.277
## LAT.cent 1 779.67 850.17 149.671
## LON.cent 1 137.96 208.46 79.386Leider kann drop1 nur den ganzen rpoly(ELEV,2) Term auf einmal entfernen und nicht, wie gewünscht nur den Term 2. Ordnung. Deswegen müssen wir dies manuell machen.
##
## Call:
## lm(formula = YEAR ~ LAT.cent + LON.cent + poly(ELEV, 1), data = temp.ru)
##
## Residuals:
## Min 1Q Median 3Q Max
## -2.46384 -0.76645 0.08496 0.77674 2.28727
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -1.98438 0.17551 -11.306 7.11e-15 ***
## LAT.cent -0.77774 0.03190 -24.378 < 2e-16 ***
## LON.cent -0.10769 0.01138 -9.467 2.26e-12 ***
## poly(ELEV, 1) -14.64838 1.46263 -10.015 3.88e-13 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.241 on 46 degrees of freedom
## Multiple R-squared: 0.9333, Adjusted R-squared: 0.929
## F-statistic: 214.7 on 3 and 46 DF, p-value: < 2.2e-16Wenn wir das mit den Koeffizienten aus dem Modell mit den Haupteffekten ohne poly vergleichen, sehen wir, das der Koeffizient für ELEV unterschiedlich ist. Wie gesagt, es sind neue Hilfsvariablen. Beim Vorhersagen mit predict kommt trotzdem das gleich heraus. Einfacher ist es aber, in diesem Fall mit dem normalen Haupteffekt Modell g zu arbeiten.
## [1] 171.0707## [1] 169.31915.2.1.2 Interaktionen
Nun wollen wir untersuchen, ob Interaktionen relevant sind. Der ^ Operator in einem formula-Objekt bewirkt, dass alle Interaktionen erster Ordnung zwischen den Prädiktoren in der Klammer berücksichtigt werden.
##
## Call:
## lm(formula = YEAR ~ (ELEV + LAT.cent + LON.cent)^2, data = temp.ru)
##
## Residuals:
## Min 1Q Median 3Q Max
## -2.7112 -0.7016 0.1724 0.7027 2.3213
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -0.8374443 0.3924021 -2.134 0.0386 *
## ELEV -0.0005087 0.0021350 -0.238 0.8128
## LAT.cent -0.7603968 0.0326131 -23.316 < 2e-16 ***
## LON.cent -0.0938562 0.0144875 -6.478 7.36e-08 ***
## ELEV:LAT.cent 0.0004796 0.0002566 1.869 0.0685 .
## ELEV:LON.cent -0.0002094 0.0001026 -2.041 0.0474 *
## LAT.cent:LON.cent -0.0014476 0.0014300 -1.012 0.3171
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.211 on 43 degrees of freedom
## Multiple R-squared: 0.9406, Adjusted R-squared: 0.9324
## F-statistic: 113.6 on 6 and 43 DF, p-value: < 2.2e-16Wir sehen jede Menge nicht signifikanter Terme. Mittels drop1 ermitteln wir, welchen Term wir als erstes (niemals mehr als einen auf einmal entfernen, da sich alle Koeffizienten, Standardfehler und Signifikanzniveaus der anderen Terme dabei verändern) entfernen sollten.
## Single term deletions
##
## Model:
## YEAR ~ (ELEV + LAT.cent + LON.cent)^2
## Df Sum of Sq RSS AIC
## <none> 63.076 25.616
## ELEV:LAT.cent 1 5.1234 68.199 27.521
## ELEV:LON.cent 1 6.1123 69.188 28.240
## LAT.cent:LON.cent 1 1.5032 64.579 24.794Dies setzen wir am einfachsten mittels update um - der : Operator bezeichnet dabei die Interaktion zwischen den beiden beteiligten Haupteffekten:
## Single term deletions
##
## Model:
## YEAR ~ ELEV + LAT.cent + LON.cent + ELEV:LAT.cent + ELEV:LON.cent
## Df Sum of Sq RSS AIC
## <none> 64.579 24.794
## ELEV:LAT.cent 1 3.6343 68.214 25.531
## ELEV:LON.cent 1 4.6515 69.231 26.271Da der \(\delta\) AIC kleiner 2 ist würde ich weitere Terme entfernen:
## Single term deletions
##
## Model:
## YEAR ~ ELEV + LAT.cent + LON.cent + ELEV:LON.cent
## Df Sum of Sq RSS AIC
## <none> 68.21 25.531
## LAT.cent 1 824.86 893.07 152.132
## ELEV:LON.cent 1 2.63 70.85 25.425## Single term deletions
##
## Model:
## YEAR ~ ELEV + LAT.cent + LON.cent
## Df Sum of Sq RSS AIC
## <none> 70.85 25.425
## ELEV 1 154.48 225.33 81.277
## LAT.cent 1 915.28 986.13 155.088
## LON.cent 1 138.03 208.88 77.487Hier sind wir am Ende angelangt.
Wir könnten den Ablauf auch über step durchführen, der drop1 und update automatisch aufruft.
## Start: AIC=25.62
## YEAR ~ (ELEV + LAT.cent + LON.cent)^2
##
## Df Sum of Sq RSS AIC
## - LAT.cent:LON.cent 1 1.5032 64.579 24.794
## <none> 63.076 25.616
## - ELEV:LAT.cent 1 5.1234 68.199 27.521
## - ELEV:LON.cent 1 6.1123 69.188 28.240
##
## Step: AIC=24.79
## YEAR ~ ELEV + LAT.cent + LON.cent + ELEV:LAT.cent + ELEV:LON.cent
##
## Df Sum of Sq RSS AIC
## <none> 64.579 24.794
## - ELEV:LAT.cent 1 3.6343 68.214 25.531
## - ELEV:LON.cent 1 4.6515 69.231 26.271##
## Call:
## lm(formula = YEAR ~ ELEV + LAT.cent + LON.cent + ELEV:LAT.cent +
## ELEV:LON.cent, data = temp.ru)
##
## Coefficients:
## (Intercept) ELEV LAT.cent LON.cent ELEV:LAT.cent
## -0.5442916 -0.0019156 -0.7647990 -0.0916829 0.0003277
## ELEV:LON.cent
## -0.0001658Allerdings hört die Funktion hier zu früh auf, da das \(\delta\) AIC nicht beachtet wird.
15.2.2 Kanada
Durch seine geographische Lage (große Wasserkörper auf der westlichen und der östlichen Seite), könnte man für Kanada einen quadratischen Effekt hinsichtlich des Längengrades auf die Jahresmitteltemperatur vermuten, ggf. auch eine Interaktio.
Laden der Daten und fitten des Modells nur für die Haupteffekte
temp.ca <- temp %>% filter(CNTRY_NAME == "Canada")
g.ca <- lm(YEAR ~ ELEV+ LAT + LON, data = temp.ca)
summary(g.ca)##
## Call:
## lm(formula = YEAR ~ ELEV + LAT + LON, data = temp.ca)
##
## Residuals:
## Min 1Q Median 3Q Max
## -3.9572 -1.1495 -0.1294 1.1152 4.3326
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 38.2020482 0.8110678 47.101 < 2e-16 ***
## ELEV -0.0016589 0.0003874 -4.282 2.75e-05 ***
## LAT -0.8760948 0.0161448 -54.265 < 2e-16 ***
## LON -0.1007708 0.0057162 -17.629 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.636 on 224 degrees of freedom
## Multiple R-squared: 0.9311, Adjusted R-squared: 0.9301
## F-statistic: 1009 on 3 and 224 DF, p-value: < 2.2e-16Test auf quadratischen Effekt
##
## Call:
## lm(formula = YEAR ~ ELEV + LAT + poly(LON, 2), data = temp.ca)
##
## Residuals:
## Min 1Q Median 3Q Max
## -3.6671 -0.7819 0.0221 0.8009 4.2127
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 4.822e+01 7.329e-01 65.794 < 2e-16 ***
## ELEV -1.186e-03 3.172e-04 -3.739 0.000235 ***
## LAT -8.862e-01 1.313e-02 -67.508 < 2e-16 ***
## poly(LON, 2)1 -3.429e+01 1.601e+00 -21.414 < 2e-16 ***
## poly(LON, 2)2 1.460e+01 1.347e+00 10.842 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.327 on 223 degrees of freedom
## Multiple R-squared: 0.9549, Adjusted R-squared: 0.9541
## F-statistic: 1179 on 4 and 223 DF, p-value: < 2.2e-16Test auf Interaktion
##
## Call:
## lm(formula = YEAR ~ ELEV + LAT * LON, data = temp.ca)
##
## Residuals:
## Min 1Q Median 3Q Max
## -3.9767 -1.0913 0.0173 1.0375 3.8072
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 26.7890324 3.5111135 7.630 6.77e-13 ***
## ELEV -0.0019757 0.0003906 -5.058 8.86e-07 ***
## LAT -0.6645984 0.0653200 -10.175 < 2e-16 ***
## LON -0.2275634 0.0384071 -5.925 1.17e-08 ***
## LAT:LON 0.0023044 0.0006906 3.337 0.000993 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.6 on 223 degrees of freedom
## Multiple R-squared: 0.9343, Adjusted R-squared: 0.9332
## F-statistic: 793.4 on 4 and 223 DF, p-value: < 2.2e-16Test auf Interaktion mit quadratischem Effekt.
##
## Call:
## lm(formula = YEAR ~ ELEV + LAT * poly(LON, 2), data = temp.ca)
##
## Residuals:
## Min 1Q Median 3Q Max
## -2.67092 -0.60768 0.06757 0.67782 2.93637
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 5.010e+01 6.043e-01 82.915 < 2e-16 ***
## ELEV -1.635e-03 2.588e-04 -6.316 1.45e-09 ***
## LAT -9.127e-01 1.067e-02 -85.582 < 2e-16 ***
## poly(LON, 2)1 -1.259e+02 9.437e+00 -13.342 < 2e-16 ***
## poly(LON, 2)2 5.534e+01 8.289e+00 6.676 1.96e-10 ***
## LAT:poly(LON, 2)1 1.635e+00 1.715e-01 9.533 < 2e-16 ***
## LAT:poly(LON, 2)2 -6.650e-01 1.501e-01 -4.430 1.48e-05 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.053 on 221 degrees of freedom
## Multiple R-squared: 0.9718, Adjusted R-squared: 0.9711
## F-statistic: 1271 on 6 and 221 DF, p-value: < 2.2e-16Vergleichen wir die Modelle einmal mittels AIC:
## [1] 877.5011## [1] 782.9726## [1] 868.3921## [1] 679.3417Das komplexe Modell wird favorisiert, da der AIC am kleinsten ist.
Um zu verstehen, wie die Zusammenhänge aussehen, müssen wir predict benutzen. Bereiten wir zunächst die Daten für die Vorhersage vor. expand.grid erstellt dabei alle möglichen Kombinationen der einzelnen Felder - wir wollen alles 3 Prädiktoren varieren. Allerdings verwenden wir für die Geländehöhe und den Längengrad nur 3 Werte, während wir für den Breitenkreis - der den stärksten Effekt hat - 100 Werte. Die Motivation liegt darin, wie wir die Zusammenhänge am Ende plotten wollen.
newdatCa <- expand.grid(ELEV = quantile(temp.ca$ELEV,
probs = c(.25, .5, .75)),
LAT = seq(min(temp.ca$LAT),
max(temp.ca$LAT), length.out = 100),
LON = quantile(temp.ca$LON,
probs = c(.25, .5, .75)))Nun wenden wir predict an.
predMain <- predict(g.ca, newdata = newdatCa, interval = "confidenc") %>%
as.data.frame()
predPoly2 <- predict(g.caPoly2, newdata = newdatCa, interval = "confidenc") %>%
as.data.frame()
predInt <- predict(g.caInt, newdata = newdatCa, interval = "confidenc") %>%
as.data.frame()
predIntPoly <- predict(g.caIntPoly, newdata = newdatCa, interval = "confidenc") %>%
as.data.frame()Die Vorhersage hängen wir für das Plotten an den data.frame mit den für die Vorhersage benutzten Werten für die Prädiktoren an.
newdatCa$predMain <- predMain$fit
newdatCa$predMain_lwr <- predMain$lwr
newdatCa$predMain_upr <- predMain$upr
newdatCa$predPoly <- predPoly2$fi
newdatCa$predPoly_lwr <- predPoly2$lwr
newdatCa$predPoly_upr <- predPoly2$upr
newdatCa$predInt <- predInt$fit
newdatCa$predInt_lwr <- predInt$lwr
newdatCa$predInt_upr <- predInt$upr
newdatCa$pred_quadIn <- predIntPoly$fit
newdatCa$pred_quadIn_lwr <- predIntPoly$lwr
newdatCa$pred_quadIn_upr <- predIntPoly$uprNun bringen wir den dat.frame vom weiten in ein langes Format für das Plotten mit ggplot2. Hierzu verwenden wir ein Pivotierungserkzeug, wie es aus der Einführung in die Geoinformatik bekannt sein dürfte.
prediction_long <- pivot_longer(newdatCa, cols = -(1:3))
idx_main <- grep(pattern = "Main", prediction_long$name)
idx_poly <- grep(pattern = "Poly", prediction_long$name)
idx_int <- grep(pattern = "Int", prediction_long$name)
idx_intpoly <- grep(pattern = "quadIn", prediction_long$name)
idx_lwr <- grep(pattern = "lwr", prediction_long$name)
idx_upr <- grep(pattern = "upr", prediction_long$name)
prediction_long$model <- "main"
prediction_long$model[idx_poly] <- "quadratic"
prediction_long$model[idx_int] <- "interaction"
prediction_long$model[idx_intpoly] <- "interaction quadratic"
prediction_long$type <- "fit"
prediction_long$type[idx_lwr] <- "lwr"
prediction_long$type[idx_upr] <- "upr"Nun der eigentliche Plot
prediction_long%>% filter(type == "fit") %>%
ggplot( mapping = aes(
x = LAT, y = value, lty = type, color = model)) +
geom_line() +
facet_wrap(~ LON + ELEV, labeller = label_both) 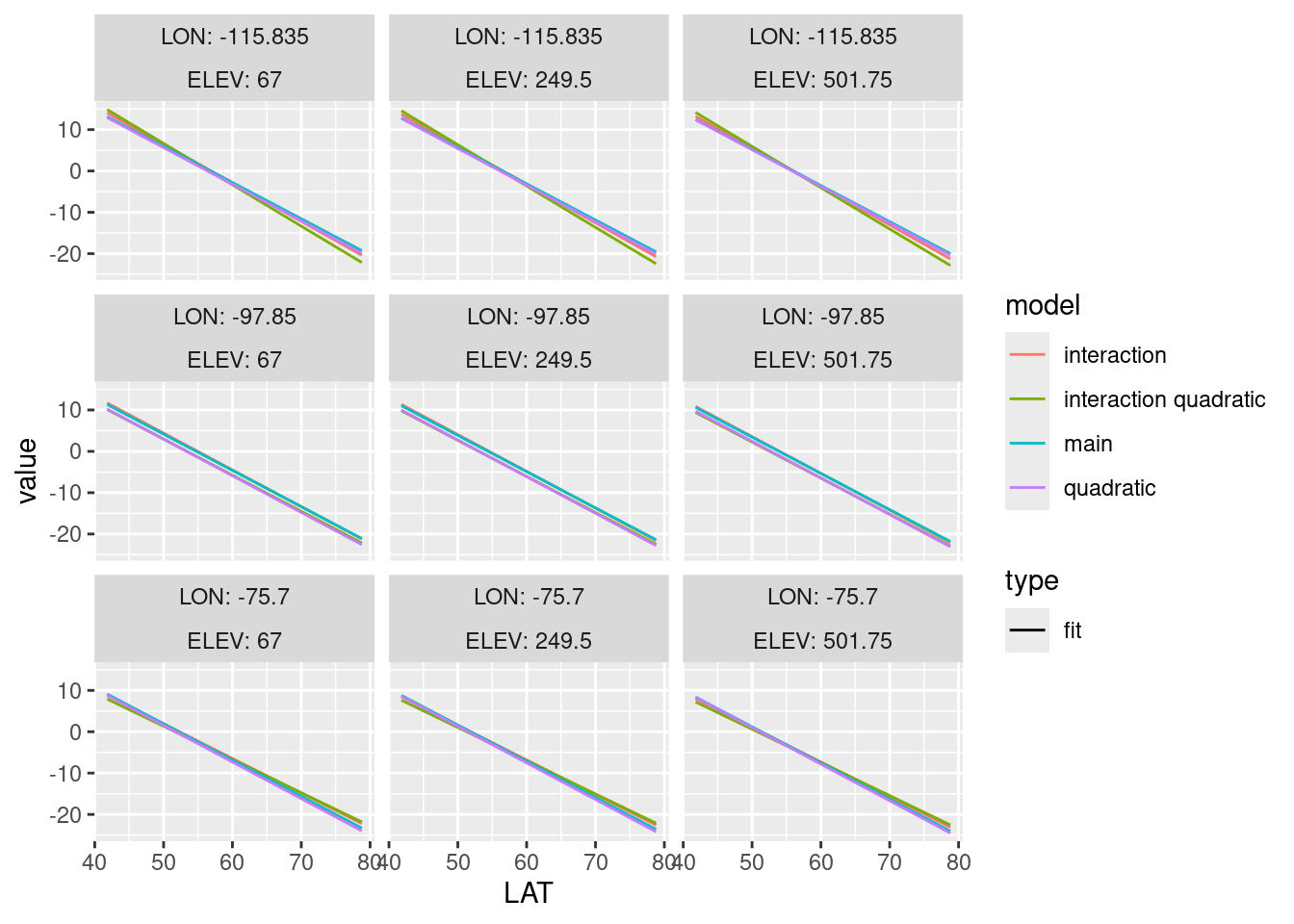
Wie wir sehen, haben wir nur sehr schwache nichtlineare Effekte im Modell. Signifikanz ist nicht das Gleiche wie Bedeutsamkeit.
15.2.3 All-subset Regression
Anstelle der stepwise regression wird oftmals die all-subset Regression empfohlen, die alle Kombinationen testet. Das Packet leaps übernimmt das für uns. Allerdings beachtet es dabei nicht das Prinzip der Marginalität und es kann nur für das lineare Modell eingesetzt werden.
Wir schauen uns dies einmal für die russischen Klimastationen an.
#####################################################
#### Using an all-subset-regression
#####################################################
require(leaps) # das Package muss vermutlich installiert werden## Loading required package: leapssubs <- regsubsets(YEAR ~ poly(ELEV,2) + poly(LAT.cent,2) + poly(LON.cent,2) , data=temp.ru, method="exhaustive", nbest=1)
(subs.s <- summary(subs))## Subset selection object
## Call: regsubsets.formula(YEAR ~ poly(ELEV, 2) + poly(LAT.cent, 2) +
## poly(LON.cent, 2), data = temp.ru, method = "exhaustive",
## nbest = 1)
## 6 Variables (and intercept)
## Forced in Forced out
## poly(ELEV, 2)1 FALSE FALSE
## poly(ELEV, 2)2 FALSE FALSE
## poly(LAT.cent, 2)1 FALSE FALSE
## poly(LAT.cent, 2)2 FALSE FALSE
## poly(LON.cent, 2)1 FALSE FALSE
## poly(LON.cent, 2)2 FALSE FALSE
## 1 subsets of each size up to 6
## Selection Algorithm: exhaustive
## poly(ELEV, 2)1 poly(ELEV, 2)2 poly(LAT.cent, 2)1 poly(LAT.cent, 2)2
## 1 ( 1 ) " " " " "*" " "
## 2 ( 1 ) "*" " " "*" " "
## 3 ( 1 ) "*" " " "*" " "
## 4 ( 1 ) "*" " " "*" "*"
## 5 ( 1 ) "*" " " "*" "*"
## 6 ( 1 ) "*" "*" "*" "*"
## poly(LON.cent, 2)1 poly(LON.cent, 2)2
## 1 ( 1 ) " " " "
## 2 ( 1 ) " " " "
## 3 ( 1 ) "*" " "
## 4 ( 1 ) "*" " "
## 5 ( 1 ) "*" "*"
## 6 ( 1 ) "*" "*"Im Plot sieht das viel übersichtlicher aus:
###################################################
### How does performance change with the number of parameters?
###################################################
par(mfrow=c(2,2), mar=c(2.7,4.7,1,1), oma=c(2,0,0,0))
plot(1:6, subs.s$rsq, xlab="# Parameter", ylab=expression(R^2), pch=21, bg="blue", cex=1.5, type="b")
plot(1:6, subs.s$adjr2, xlab="# Parameter", ylab=expression(paste(sep="", R^2, ".adj")), pch=21, bg="blue", cex=1.5, type="b")
plot(1:6, subs.s$bic, xlab="# Parameter", ylab="BIC", pch=21, bg="blue", cex=1.5, type="b")
plot(1:6, subs.s$rss, xlab="# Parameter", ylab="RSS", pch=21, bg="blue", cex=1.5, type="b")
mtext(text = "# parameters", side = 1, line = 3, at = 0)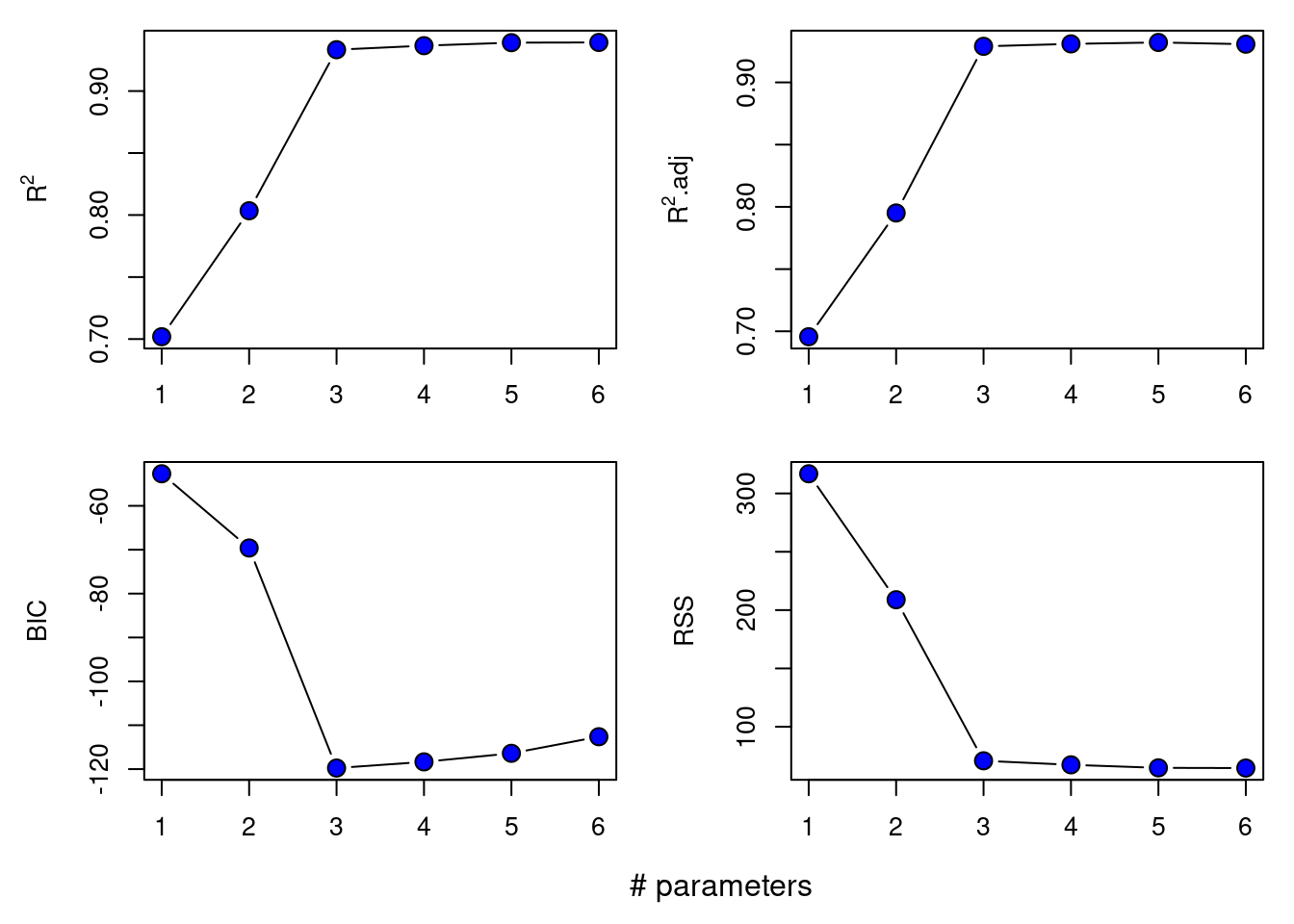
Das Modell mit 3 Parameter wird vom BIC favorisiert.
Nun wollen wir aber auch wissen, welche Variablen dabei im Modell sind:
###################################################
### which variables are in models with 1,2,3,... variables
### and how does the goodness-of-fit indicator react?
###################################################
par(mfrow=c(1,3), oma=c(3,0,0,0), mar=c(8.1, 1.1, .1,.1), cex.lab=1.4)
plot(subs)
plot(subs, scale="adjr2")
plot(subs, scale="r2")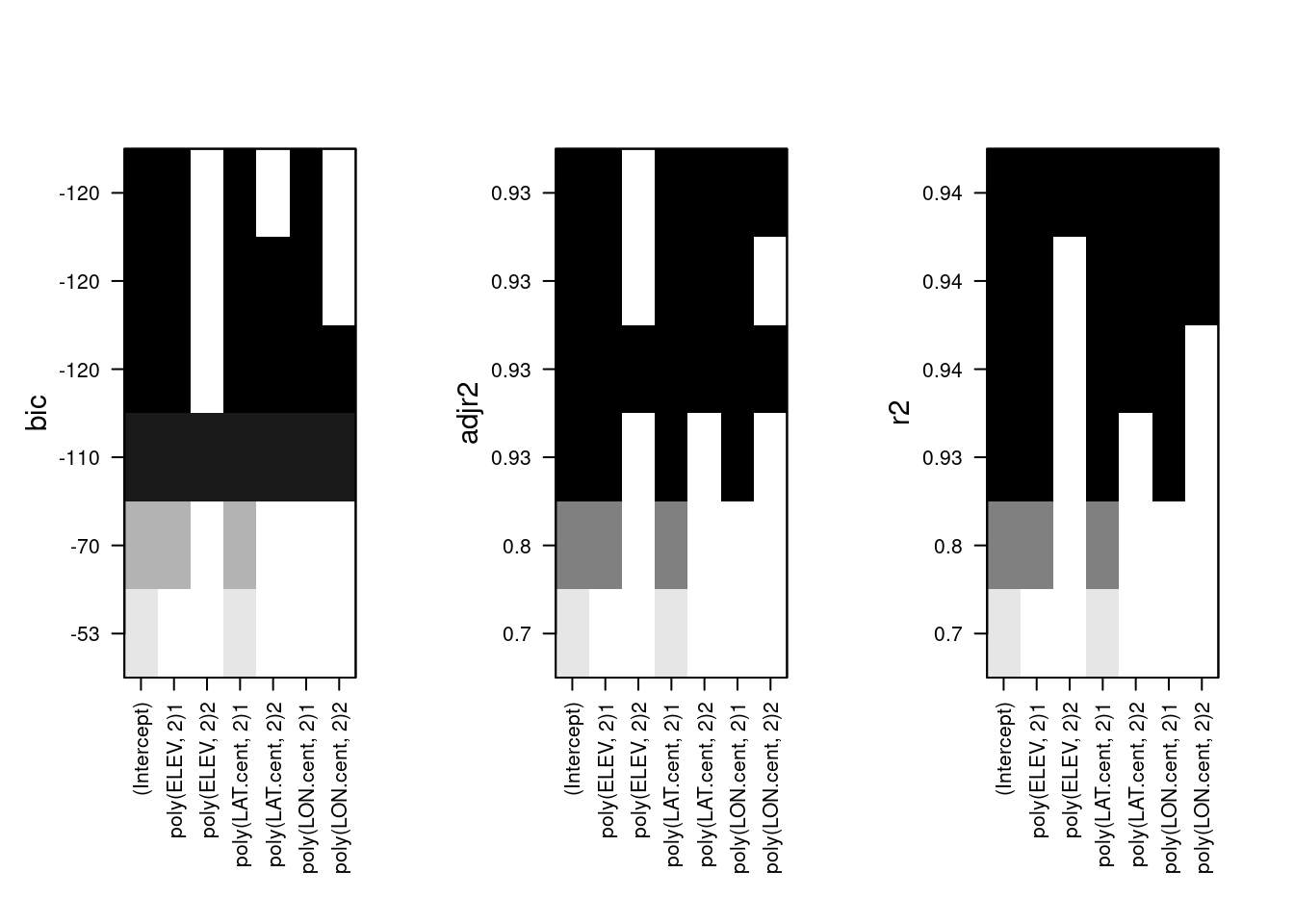
Wir können auch die Koeffizienten extrahieren - hier für die Modelle mit 1 bis 4 Parametern:
## [[1]]
## (Intercept) poly(LAT.cent, 2)1
## -1.984379 -27.311679
##
## [[2]]
## (Intercept) poly(ELEV, 2)1 poly(LAT.cent, 2)1
## -1.984379 -12.023801 -33.362243
##
## [[3]]
## (Intercept) poly(ELEV, 2)1 poly(LAT.cent, 2)1 poly(LON.cent, 2)1
## -1.984379 -14.648379 -43.173925 -14.672258
##
## [[4]]
## (Intercept) poly(ELEV, 2)1 poly(LAT.cent, 2)1 poly(LAT.cent, 2)2
## -1.984379 -14.532479 -42.087418 -2.348418
## poly(LON.cent, 2)1
## -12.895568Für das favorisierte 3 Parameter Modell sind wir also beim ursprünglichen Modell mit linearen Termen für Geländehöhe, Breitengrad und Längengrad.
Oder auch die Varianz-Covarianz-Matrix für die Regressionskoeffizienten ausgeben lassen - hier für das 1-Parameter Modell:
## (Intercept) poly(LAT.cent, 2)1
## (Intercept) 0.1320185 0.000000
## poly(LAT.cent, 2)1 0.0000000 6.60092615.3 Anwendungsbeispiel CO2 Konzentrationen
Wir laden die athmosphärischen in situ Messungen der \(\textrm{CO}_2\) Konzentration am Standort Mauna Loa, Hawaii (Thoning, Crotwell, and Mund 2020), die bereits vorprozessiert und als Rdata Datei abgespeichert wurden 74. Der loadBefehl lädt die Rdata Datei und macht die gespeicherten (serialisierten) Objekte wieder verfügbar - hier den data.frame co2InSitu.
Die Daten zeigen die bekannte Zunahme der athmosphärischen \(\textrm{CO}_2\) zusätzlich zu den - für diesen Breitengrad - typischen saisonalen Schankungen aufgrund der Vegetationsentwicklung auf.
ggplot(co2InSitu, mapping = aes(x=Date, y=value)) + geom_line() +
labs(title="C02 in situ Messungen", subtitle = "Mauna Loa, Hawaii, United States") +
xlab("") + ylab("CO2 [ppm]")## Warning: Removed 501 rows containing missing values or values outside the scale range
## (`geom_line()`).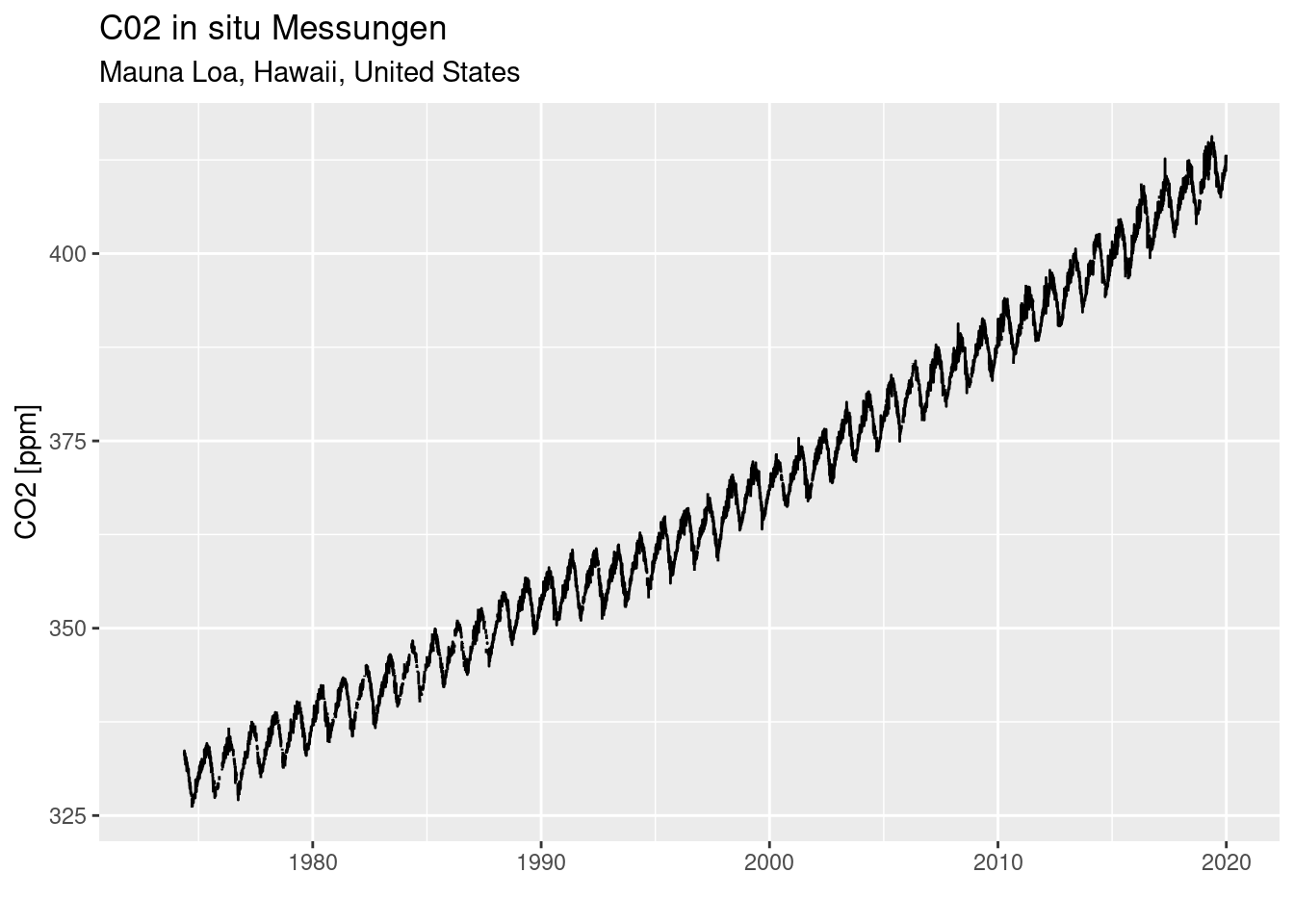
Wir können die Zunahme mit einem Polynom ersten oder zweiten Grades modellierten.
Beide Modell unterscheiden sich nur unwesentlich hinsichtlich des adjustierten \(R^2\) Wertes. Sind sie trotzdem unterschiedlich hinsichtlich der Modelgüte?
##
## Call:
## lm(formula = value ~ Date, data = co2InSitu)
##
## Residuals:
## Min 1Q Median 3Q Max
## -8.5595 -2.2804 -0.0345 2.1810 10.4171
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 3.182e+02 6.380e-02 4986.9 <2e-16 ***
## Date 5.631e-08 6.533e-11 861.8 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 3.149 on 13944 degrees of freedom
## (3220 observations deleted due to missingness)
## Multiple R-squared: 0.9816, Adjusted R-squared: 0.9816
## F-statistic: 7.427e+05 on 1 and 13944 DF, p-value: < 2.2e-16##
## Call:
## lm(formula = value ~ poly(Date, 2), data = co2InSitu)
##
## Residuals:
## Min 1Q Median 3Q Max
## -6.580 -1.879 0.095 1.855 7.463
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 3.655e+02 2.043e-02 17886.7 <2e-16 ***
## poly(Date, 2)1 3.108e+03 2.812e+00 1105.3 <2e-16 ***
## poly(Date, 2)2 2.863e+02 2.796e+00 102.4 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 2.38 on 13943 degrees of freedom
## (3220 observations deleted due to missingness)
## Multiple R-squared: 0.9895, Adjusted R-squared: 0.9895
## F-statistic: 6.557e+05 on 2 and 13943 DF, p-value: < 2.2e-16Ein Modellvergleich mittels AIC zeigt klar, dass das Modell mit dem Polynom zweiten Grades den Verlauf deutlich besser beschreibt.
## [1] 71578.22## [1] 63761.67Ein likelihood-ratio-Test kommt zum gleichen klaren Ergebnis.
## Likelihood ratio test
##
## Model 1: value ~ Date
## Model 2: value ~ poly(Date, 2)
## #Df LogLik Df Chisq Pr(>Chisq)
## 1 3 -35786
## 2 4 -31877 1 7818.6 < 2.2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Ein visueller Vergleich (Abbildung 15.4) zeigt ebenfalls, dass das Modell mit dem Polynom 2. Grades den Trend besser beschreibt als ein einfacher linearer Zusammenhang. Insbesondere trifft die Vorhersage die Werte zum Ende der Beobachtungsperiode besser als der einfache lineare Zusammenhang.
p1 <- ggplot(co2InSitu, mapping = aes(x=Date, y=value)) + geom_line() +
geom_smooth(method="lm", col="red") +
labs(title="C02 in situ Messungen", subtitle = "Mauna Loa, Hawaii, United States - Poly1") +
xlab("") + ylab("CO2 [ppm]")
p2 <- ggplot(co2InSitu, mapping = aes(x=Date, y=value)) + geom_line() +
geom_smooth(method="lm", formula= y ~poly(x,2), col="red") +
labs(title="C02 in situ Messungen", subtitle = "Mauna Loa, Hawaii, United States - Poly 2") +
xlab("") + ylab("CO2 [ppm]")
ggarrange(p1,p2)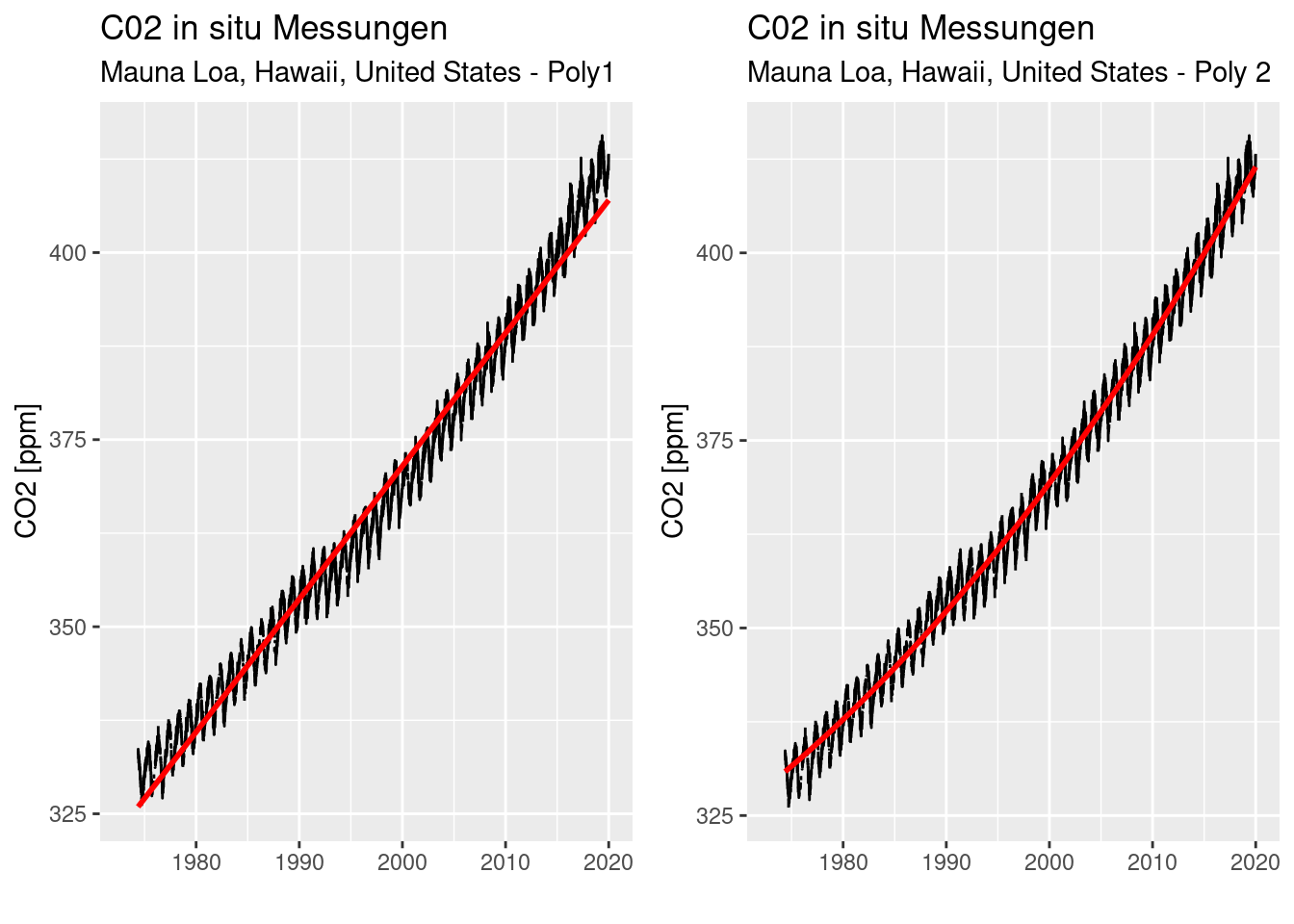
Figure 15.4: Vergleich eines inearen (links) und eines quadratischen (rechts) Vorhersagemodells für die athmosphärischen CO2 Konzentrationen am Standort Mauna Loa, Hawaii.
15.4 Anwendungsbeispiel Baumdichte und Klima
Bisher waren die Nichtlinearen Zusammenhänge eher nicht so überzeugend. Deswegen hier ein Beispiel, bei dem die Effekte bedeutsam sind.
Interaktionen und polynome Terme testen wir anhand eines Datensatzes von Anadón, Sala, and Maestre (2014). Der Datensatz enthält Informationen zur Baumdichte in Lateinamerika sowie klimatische Prädiktoren.
## X Y BIO1 BIO12
## Min. :-176.55 Min. :-55.141 Min. :-273.24 Min. : 0.0
## 1st Qu.:-108.89 1st Qu.: -4.733 1st Qu.: -44.00 1st Qu.: 309.3
## Median : -85.09 Median : 41.384 Median : 70.90 Median : 611.4
## Mean : -89.17 Mean : 29.502 Mean : 71.95 Mean : 876.5
## 3rd Qu.: -66.85 3rd Qu.: 59.887 3rd Qu.: 209.00 3rd Qu.:1225.6
## Max. : -34.98 Max. : 83.013 Max. : 288.00 Max. :9431.2
## PET MOD44B GLC
## Min. : 49.96 Min. : 0.00 Min. : 1.000
## 1st Qu.: 477.40 1st Qu.: 0.00 1st Qu.: 4.000
## Median : 982.40 Median : 3.58 Median :12.000
## Mean :1002.31 Mean :17.81 Mean : 9.639
## 3rd Qu.:1578.20 3rd Qu.:27.86 3rd Qu.:14.000
## Max. :2078.52 Max. :84.05 Max. :22.000Die Daten liegen in einem regelmäßigen Raster vor. Folgende Attributwerte sind für uns im weiteren relevant.
- MOD44B enthält den aus MODIS Fernerkundungsdaten abgeleiteten Flächendeckungsgrad von Bäumen (Tree cover [%*100]) je Rasterzelle.
- BIO1 - enthält die mittlere Jahrestemperatur der Zelle (Annual Mean Temperature)
- BIO12 - enthält die Jahressumme der Niederschläge in der Zelle (Annual Precipitation)
- GLC - sind Landnutzungsdaten aufgrund der Global land cover Datensatzes.
Wir wollen uns hier mit dem Zusammenhang zwischen Baumbedeckungsgrad und Klimavariablen in allen Zellen beschäftigen, die zu LN-Klasse 2 (GLC == 2: Tree cover, broadleaved,deciduous, closed, Closed cover > 40%) gehören.
Die Selektion führe ich mittels des subset durch.
Bevor wir uns an die Analyse machen, teilen wir die Daten in einen Trainings- und einen Validierungsdatensatz auf. Damit können wir eine bessere Vorhersage hinsichtlich des Vorhersagefehlers auf unbekannten Daten erhalten (Generalisierungsfehler).
Damit wir immer dieselben Aufteilung verwenden setze ich den Zufallszahlengenerator auf einen fixen Wert
set.seed(42)
n <- nrow(dat.glc2)
idx_test <- sample(x = 1:n, size = 0.2*n, replace = FALSE)
dat.glc2_test <- dat.glc2[idx_test,]
dat.glc2_train <- dat.glc2[-idx_test,]
dim(dat.glc2_test)## [1] 108 7## [1] 432 715.4.1 Explorative Analyse
pairs(dat.glc2_train[, c("MOD44B", "BIO1", "BIO12")], lower.panel = panel.smooth, diag.panel = panel.hist, upper.panel = panel.cor, labels = c("Baumbedeckung", "Temperatur", "Niederschlag"), las=1)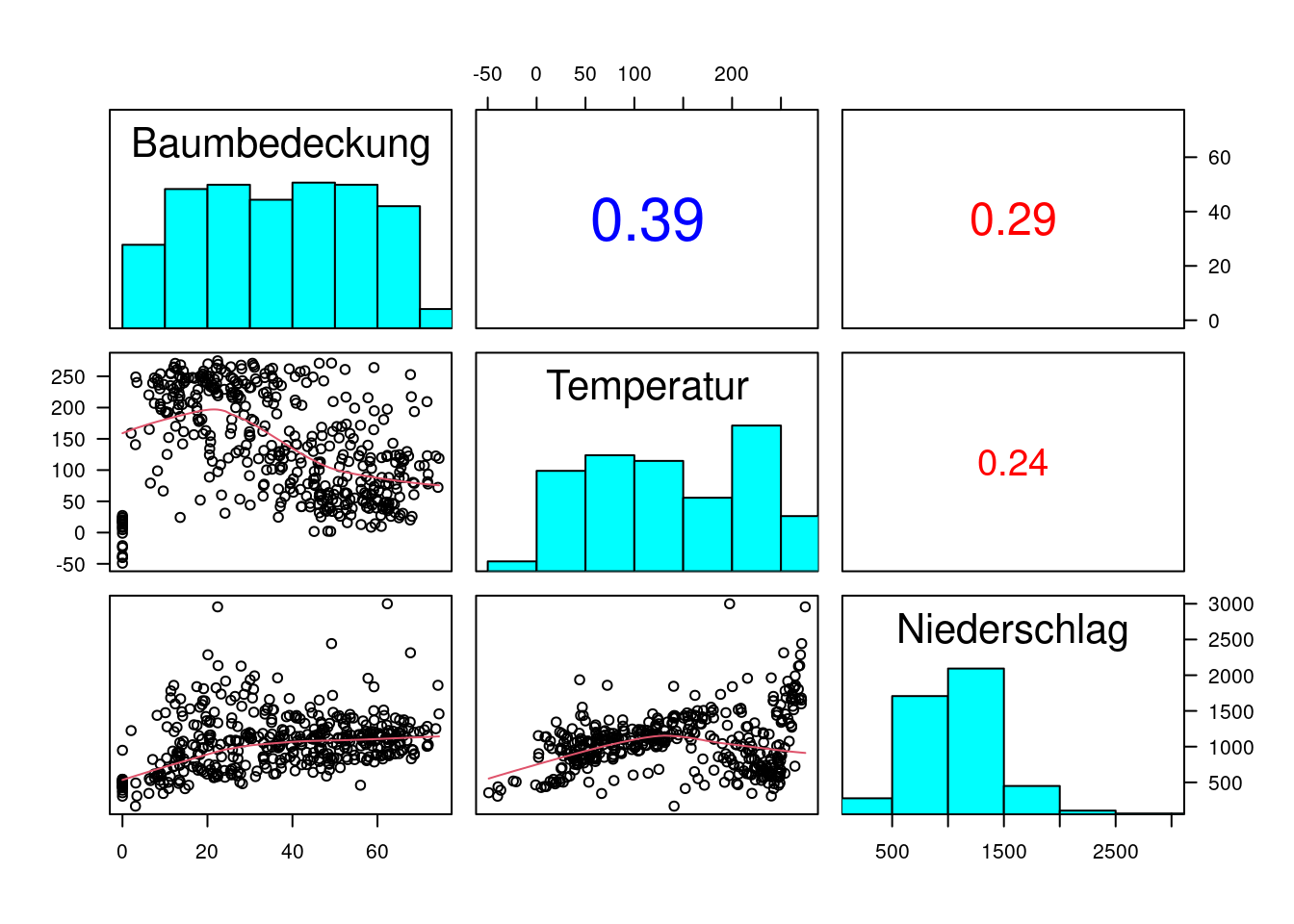
15.4.2 Haupteffekte
##
## Call:
## lm(formula = MOD44B ~ BIO1 + BIO12, data = dat.glc2_train)
##
## Residuals:
## Min 1Q Median 3Q Max
## -47.946 -9.491 1.474 11.593 44.659
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 31.161520 2.463819 12.648 <2e-16 ***
## BIO1 -0.114719 0.009870 -11.622 <2e-16 ***
## BIO12 0.020586 0.002109 9.763 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 16.55 on 429 degrees of freedom
## Multiple R-squared: 0.3034, Adjusted R-squared: 0.3002
## F-statistic: 93.43 on 2 and 429 DF, p-value: < 2.2e-16Wir sehen, dass der Baumbedeckungsgrad mit zunehmender Temperatur abnimmt und mit zunehmendem Niederschlag zunimmt.
Plotten wir das in der üblichen Form.
Zunächst erstelle ich insgesamt sechs data.frames in dem ich jeweils Temperatur oder Niederschlag festhalte und den anderen Wert auf einem konstanten Wert halte.
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 167.3 820.5 1058.6 1061.7 1241.3 3000.1## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -49.0 63.7 129.8 139.5 220.2 274.9newD.prcp <- data.frame(BIO1 = mean(dat.glc2_train$BIO1), BIO12 = seq(min(dat.glc2_train$BIO12), max(dat.glc2_train$BIO12), length.out=100))
newD.prcp.t60 <- data.frame(BIO1 = 60, BIO12 = seq(min(dat.glc2_train$BIO12), max(dat.glc2_train$BIO12), length.out=100))
newD.prcp.t250 <- data.frame(BIO1 = 250, BIO12 = seq(min(dat.glc2_train$BIO12), max(dat.glc2_train$BIO12), length.out=100))
newD.temp <- data.frame(BIO12 = mean(dat.glc2_train$BIO12), BIO1 = seq(min(dat.glc2_train$BIO1), max(dat.glc2_train$BIO1), length.out=100))
newD.temp.p600 <- data.frame(BIO12 = 600, BIO1 = seq(min(dat.glc2_train$BIO1), max(dat.glc2_train$BIO1), length.out=100))
newD.temp.p1400 <- data.frame(BIO12 = 1400, BIO1 = seq(min(dat.glc2_train$BIO1), max(dat.glc2_train$BIO1), length.out=100))Dann die Vorhersage für die sechs data.frames.
pred.prcp.main <- as.data.frame(predict(g.main, newdata = newD.prcp, interval="confidence"))
pred.prcp.t60.main <- as.data.frame(predict(g.main, newdata = newD.prcp.t60, interval="confidence"))
pred.prcp.t250.main <- as.data.frame(predict(g.main, newdata = newD.prcp.t250, interval="confidence"))
pred.temp.main <- as.data.frame(predict(g.main, newdata = newD.temp, interval="confidence"))
pred.temp.p600.main <- as.data.frame(predict(g.main, newdata = newD.temp.p600, interval="confidence"))
pred.temp.p1400.main <- as.data.frame(predict(g.main, newdata = newD.temp.p1400, interval="confidence"))Dann plotte ich die jeweils die drei Regressionslinien für konstanten Niederschlag.
plot(MOD44B ~ BIO1, data=dat.glc2_train, las=1, pch=16, col=rgb(0,0,1,.25), xlab="Jahresmitteltemperatur [°C/10]", ylab="Baumbedeckungsgrad [%]", xlim=c(-52, 400), main="Haupteffekte", cex=.1+ sqrt( BIO12)/20)
lines(pred.temp.main$fit ~ newD.temp$BIO1, col="red", lwd=2)
lines(pred.temp.main$upr ~ newD.temp$BIO1, col="red", lty=2, lwd=2)
lines(pred.temp.main$lwr ~ newD.temp$BIO1, col="red", lty=2, lwd=2)
lines(pred.temp.p600.main$fit ~ newD.temp$BIO1, col="orange", lwd=2)
lines(pred.temp.p600.main$upr ~ newD.temp$BIO1, col="orange", lty=2, lwd=2)
lines(pred.temp.p600.main$lwr ~ newD.temp$BIO1, col="orange", lty=2, lwd=2)
lines(pred.temp.p1400.main$fit ~ newD.temp$BIO1, col="firebrick4", lwd=2)
lines(pred.temp.p1400.main$upr ~ newD.temp$BIO1, col="firebrick4", lty=2, lwd=2)
lines(pred.temp.p1400.main$lwr ~ newD.temp$BIO1, col="firebrick4", lty=2, lwd=2)
legend(x="right", legend=c("600", "1057.1", "1400"), title = "Jahresniederschlag", col=c("orange", "red", "firebrick4"), lwd=2, bty='n')
prcpVals <- c(600, 1000, 1400)
legend(x=320, y=28, legend=paste(prcpVals, "mm"), pt.cex=.1+ sqrt(prcpVals)/20, pch=16, col="blue", bty='n')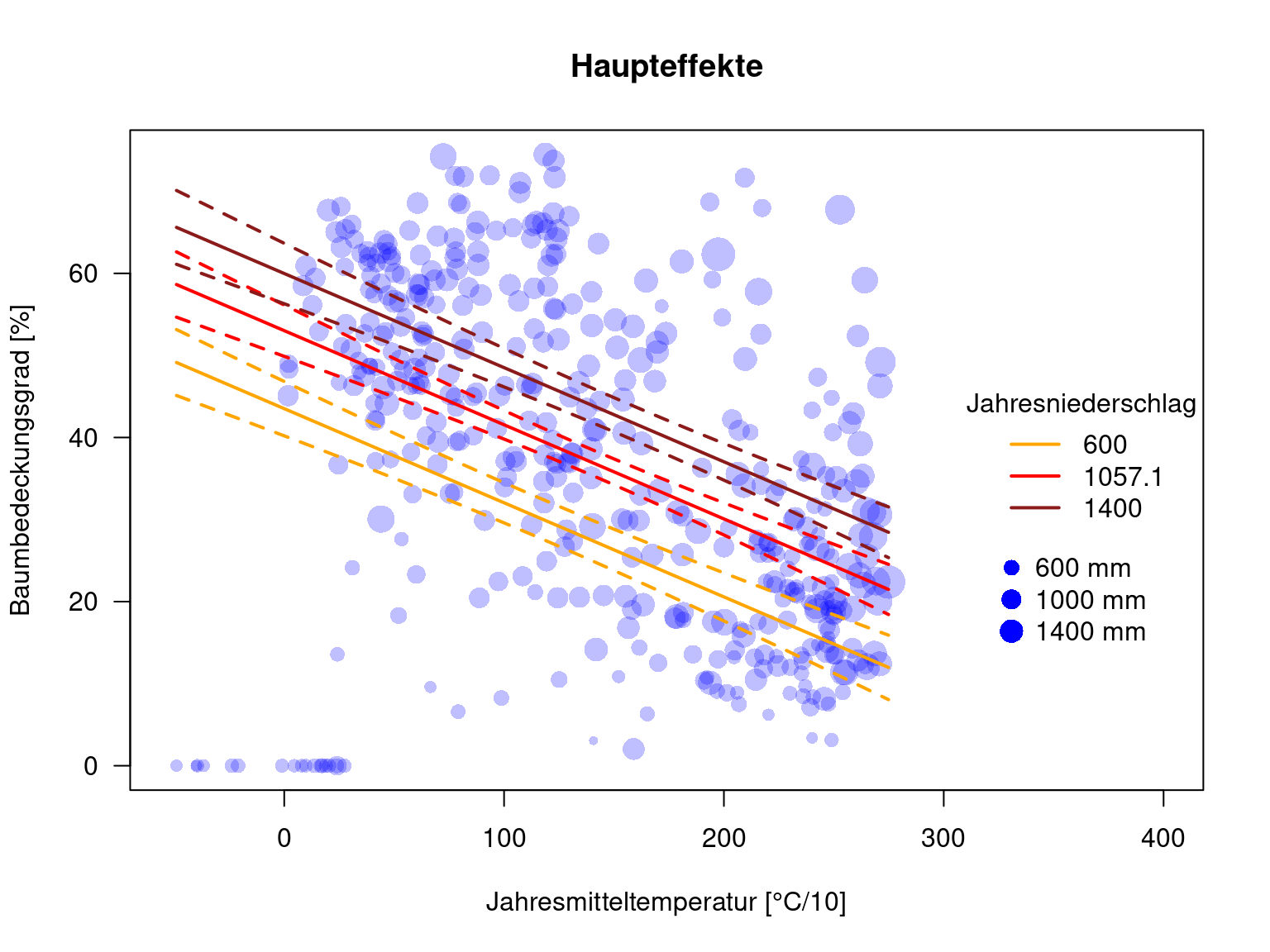
Und nun halten wir die Temperatur konstant und variieren den Niederschlag für den Plot.
plot(MOD44B ~ BIO12, data=dat.glc2_train, las=1, pch=16, col=rgb(0,0,1,.25), xlab="Jahresniederschlag [mm]", ylab="Baumbedeckungsgrad [%]", xlim=c(0, 4000), main="Haupteffekte", cex=.1+ sqrt(52+BIO1)/10)
lines(pred.prcp.main$fit ~ newD.prcp$BIO12, col="red", lwd=2)
lines(pred.prcp.main$upr ~ newD.prcp$BIO12, col="red", lty=2, lwd=2)
lines(pred.prcp.main$lwr ~ newD.prcp$BIO12, col="red", lty=2, lwd=2)
lines(pred.prcp.t60.main$fit ~ newD.prcp$BIO12, col="orange", lwd=2)
lines(pred.prcp.t60.main$upr ~ newD.prcp$BIO12, col="orange", lty=2, lwd=2)
lines(pred.prcp.t60.main$lwr ~ newD.prcp$BIO12, col="orange", lty=2, lwd=2)
lines(pred.prcp.t250.main$fit ~ newD.prcp$BIO12, col="firebrick4", lwd=2)
lines(pred.prcp.t250.main$upr ~ newD.prcp$BIO12, col="firebrick4", lty=2, lwd=2)
lines(pred.prcp.t250.main$lwr ~ newD.prcp$BIO12, col="firebrick4", lty=2, lwd=2)
legend(x=3200, y=60, legend=c("6", "14", "25"), title = "Jahresmittel-\ntemperatur [°C]", col=c("orange", "red", "firebrick4"), lwd=2, bty='n')
tempVals <- seq(-5, 30, by=10)
legend(x=3400, y=30, legend=paste(tempVals, "°C"), pt.cex=.1+ sqrt(52+tempVals*10)/10, pch=16, col="blue", bty='n')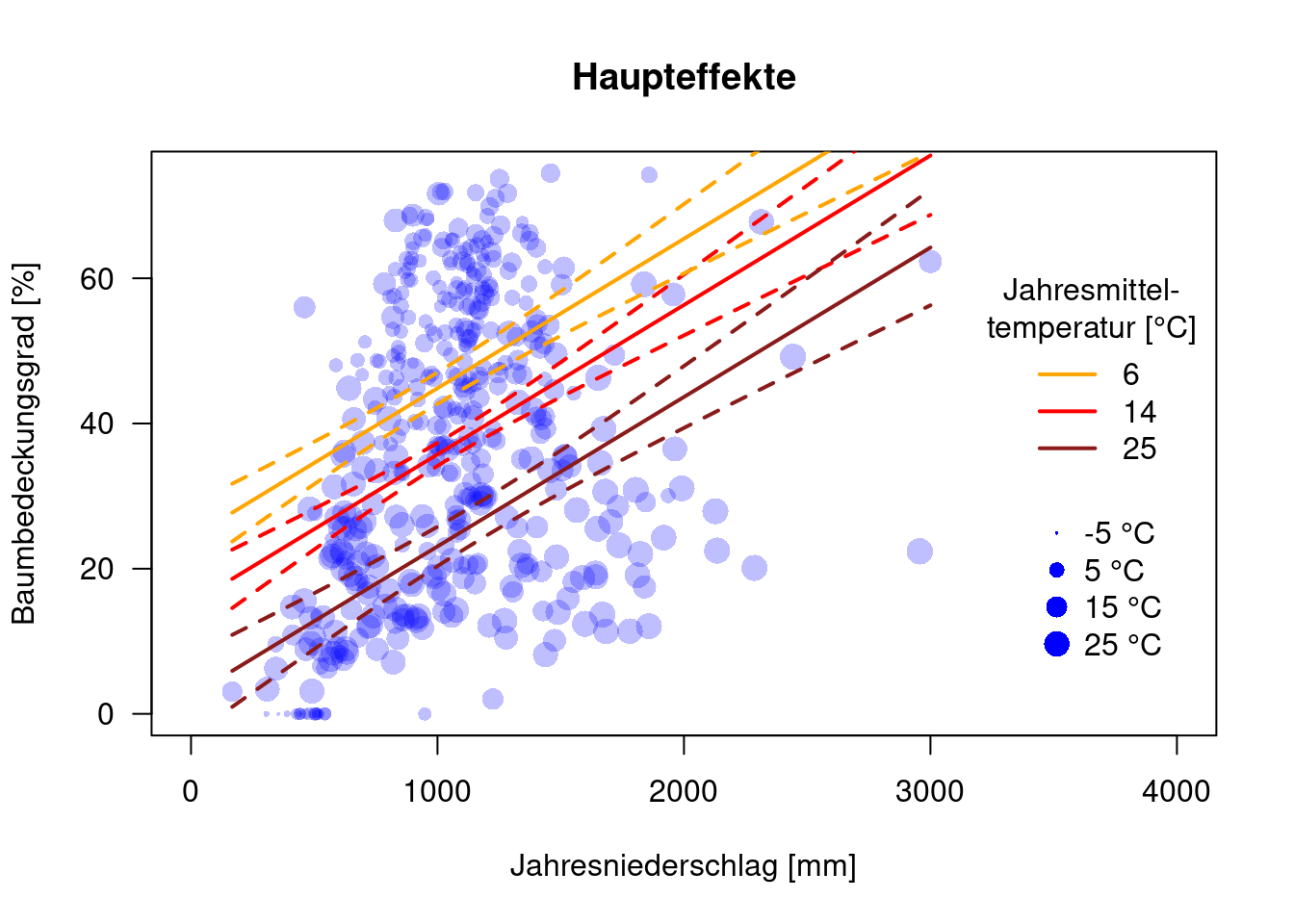
15.4.3 Interaktionen
Gibt es Wechselwirkungen zwischen Temperatur und Niederschlag?
##
## Call:
## lm(formula = MOD44B ~ BIO1 * BIO12, data = dat.glc2_train)
##
## Residuals:
## Min 1Q Median 3Q Max
## -68.367 -9.909 -0.632 9.905 43.315
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -9.620e+00 4.321e+00 -2.226 0.0265 *
## BIO1 1.033e-01 2.178e-02 4.745 2.84e-06 ***
## BIO12 6.337e-02 4.336e-03 14.615 < 2e-16 ***
## BIO1:BIO12 -2.250e-04 2.058e-05 -10.932 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 14.65 on 428 degrees of freedom
## Multiple R-squared: 0.4555, Adjusted R-squared: 0.4516
## F-statistic: 119.3 on 3 and 428 DF, p-value: < 2.2e-16Ja. Temperatur hat einen positiven Effekt auf Baumbedeckungsgrad, ebenso Niederschlag. Mit zunehmender Temperatur wird allerdings der Effekt von Niederschlag negativ (und umgekehrt).
Um das Modellverhalten besser verstehen zu können verwenden wir wieder predict und plotten dann wieder das Modellverhalten.
pred.prcp.int <- as.data.frame(predict(g.int, newdata = newD.prcp, interval="confidence"))
pred.prcp.t60.int <- as.data.frame(predict(g.int, newdata = newD.prcp.t60, interval="confidence"))
pred.prcp.t250.int <- as.data.frame(predict(g.int, newdata = newD.prcp.t250, interval="confidence"))
pred.temp.int <- as.data.frame(predict(g.int, newdata = newD.temp, interval="confidence"))
pred.temp.p600.int <- as.data.frame(predict(g.int, newdata = newD.temp.p600, interval="confidence"))
pred.temp.p1400.int <- as.data.frame(predict(g.int, newdata = newD.temp.p1400, interval="confidence"))plot(MOD44B ~ BIO1, data=dat.glc2_train, las=1, pch=16, col=rgb(0,0,1,.25), xlab="Jahresmitteltemperatur [°C/10]", ylab="Baumbedeckungsgrad [%]", xlim=c(-52, 400), main="Interaktionen", cex=.1+ sqrt( BIO12)/20)
lines(pred.temp.int$fit ~ newD.temp$BIO1, col="red", lwd=2)
lines(pred.temp.int$upr ~ newD.temp$BIO1, col="red", lty=2, lwd=2)
lines(pred.temp.int$lwr ~ newD.temp$BIO1, col="red", lty=2, lwd=2)
lines(pred.temp.p600.int$fit ~ newD.temp$BIO1, col="orange", lwd=2)
lines(pred.temp.p600.int$upr ~ newD.temp$BIO1, col="orange", lty=2, lwd=2)
lines(pred.temp.p600.int$lwr ~ newD.temp$BIO1, col="orange", lty=2, lwd=2)
lines(pred.temp.p1400.int$fit ~ newD.temp$BIO1, col="firebrick4", lwd=2)
lines(pred.temp.p1400.int$upr ~ newD.temp$BIO1, col="firebrick4", lty=2, lwd=2)
lines(pred.temp.p1400.int$lwr ~ newD.temp$BIO1, col="firebrick4", lty=2, lwd=2)
legend(x="right", legend=c("600", "1057.1", "1400"), title = "Jahresniederschlag", col=c("orange", "red", "firebrick4"), lwd=2, bty='n')
legend(x=320, y=28, legend=paste(prcpVals, "mm"), pt.cex=.1+ sqrt(prcpVals)/20, pch=16, col="blue", bty='n')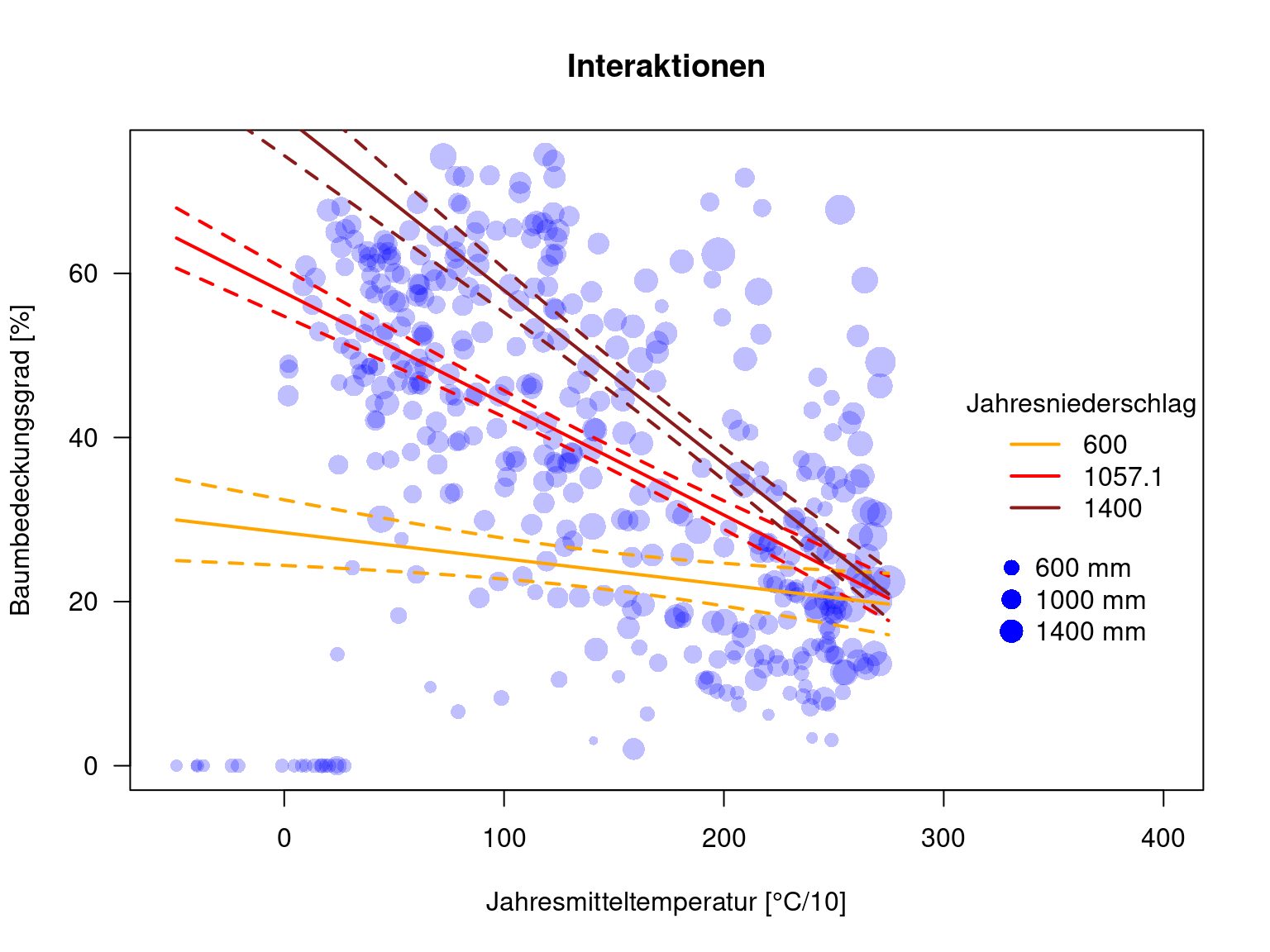
Wie wir sehen verändert sich nicht nur der offset, sondern auch die Steigung der Regressionslinie für Baumbedeckung gegen Temperatur mit dem Jahresniederschlag. Höhere Jahresniederschläge haben einen positiven Effekt auf den Baumbedeckungsgrad, allerdings sinkt der Bedeckungsgrad zugleich stärker bei zunehmender Temperatur.
plot(MOD44B ~ BIO12, data=dat.glc2_train, las=1, pch=16, col=rgb(0,0,1,.25), xlab="Jahresniederschlag [mm]", ylab="Baumbedeckungsgrad [%]", xlim=c(0, 4000), main="Interaktion", cex=.1+ sqrt(52+BIO1)/10)
lines(pred.prcp.int$fit ~ newD.prcp$BIO12, col="red", lwd=2)
lines(pred.prcp.int$upr ~ newD.prcp$BIO12, col="red", lty=2, lwd=2)
lines(pred.prcp.int$lwr ~ newD.prcp$BIO12, col="red", lty=2, lwd=2)
lines(pred.prcp.t60.int$fit ~ newD.prcp$BIO12, col="orange", lwd=2)
lines(pred.prcp.t60.int$upr ~ newD.prcp$BIO12, col="orange", lty=2, lwd=2)
lines(pred.prcp.t60.int$lwr ~ newD.prcp$BIO12, col="orange", lty=2, lwd=2)
lines(pred.prcp.t250.int$fit ~ newD.prcp$BIO12, col="firebrick4", lwd=2)
lines(pred.prcp.t250.int$upr ~ newD.prcp$BIO12, col="firebrick4", lty=2, lwd=2)
lines(pred.prcp.t250.int$lwr ~ newD.prcp$BIO12, col="firebrick4", lty=2, lwd=2)
legend(x="right", legend=c("6", "14", "25"), title = "Jahresmittel-\ntemperatur [°C]", col=c("orange", "red", "firebrick4"), lwd=2, bty='n')
tempVals <- seq(-5, 30, by=10)
legend(x=3400, y=30, legend=paste(tempVals, "°C"), pt.cex=.1+ sqrt(52+tempVals*10)/10, pch=16, col="blue", bty='n')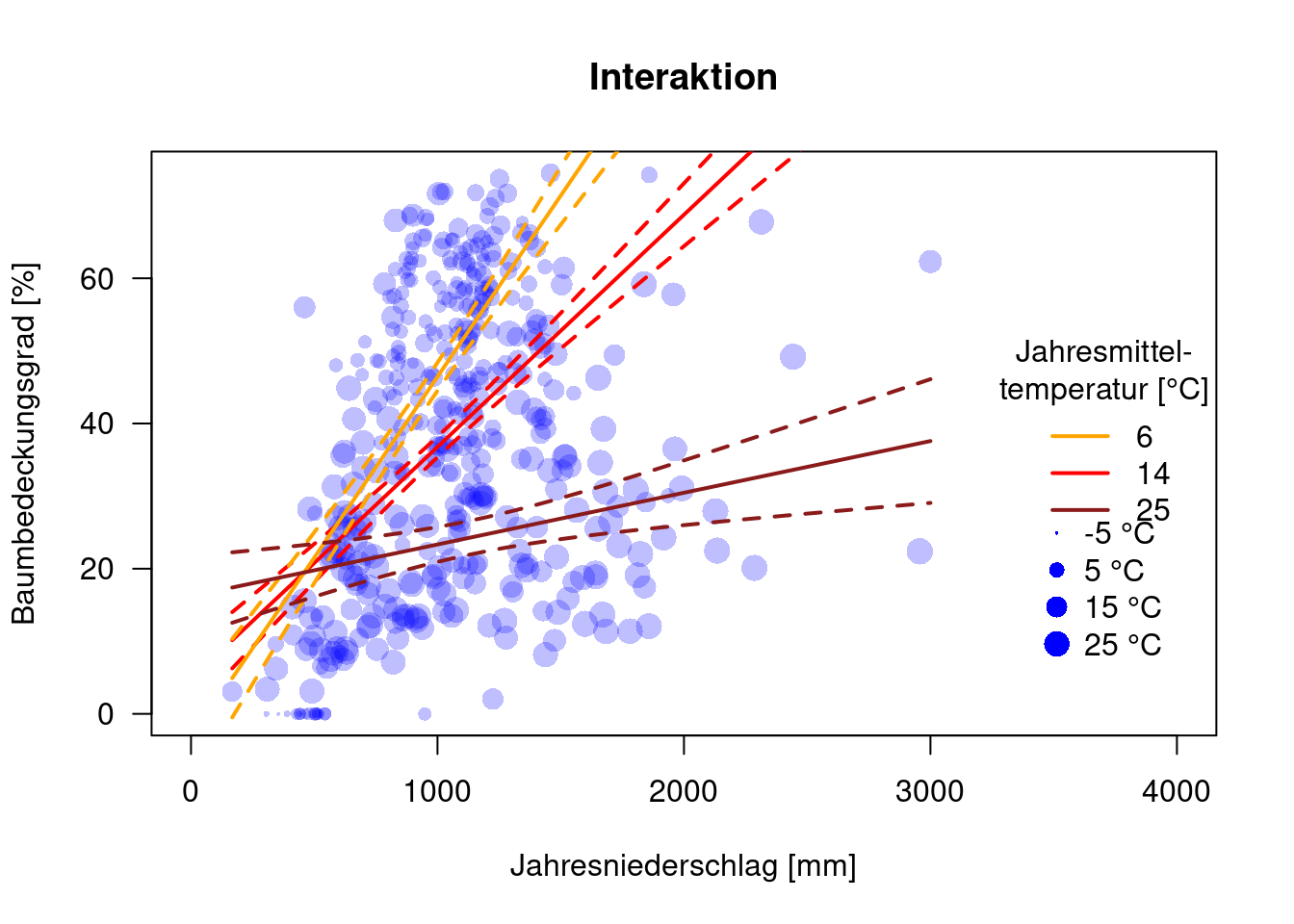
15.4.4 Polynomiale Terme
Polynomiale Terme erzeugen wir mittels poly. Der Parameter gibt an, welchen Grades das Polynom sein soll. Wenn wir von einer optimalen Klimanische ausgehen, wären quadratische Terme sinnvoll.
##
## Call:
## lm(formula = MOD44B ~ poly(BIO1, 2) + poly(BIO12, 2), data = dat.glc2_train)
##
## Residuals:
## Min 1Q Median 3Q Max
## -44.216 -10.083 0.272 10.699 42.868
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 37.014 0.735 50.361 < 2e-16 ***
## poly(BIO1, 2)1 -180.976 15.999 -11.312 < 2e-16 ***
## poly(BIO1, 2)2 -79.535 16.666 -4.772 2.50e-06 ***
## poly(BIO12, 2)1 154.043 15.822 9.736 < 2e-16 ***
## poly(BIO12, 2)2 -82.730 16.834 -4.915 1.27e-06 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 15.28 on 427 degrees of freedom
## Multiple R-squared: 0.4094, Adjusted R-squared: 0.4038
## F-statistic: 73.99 on 4 and 427 DF, p-value: < 2.2e-16Alle Terme sind signifikant.
Vorhersage mit den bereits vordefinierten data frames.
pred.prcp.poly <- as.data.frame(predict(g.poly, newdata = newD.prcp, interval="confidence"))
pred.prcp.t60.poly <- as.data.frame(predict(g.poly, newdata = newD.prcp.t60, interval="confidence"))
pred.prcp.t250.poly <- as.data.frame(predict(g.poly, newdata = newD.prcp.t250, interval="confidence"))
pred.temp.poly <- as.data.frame(predict(g.poly, newdata = newD.temp, interval="confidence"))
pred.temp.p600.poly <- as.data.frame(predict(g.poly, newdata = newD.temp.p600, interval="confidence"))
pred.temp.p1400.poly <- as.data.frame(predict(g.poly, newdata = newD.temp.p1400, interval="confidence"))Plotten der Zusammenhänge
plot(MOD44B ~ BIO1, data=dat.glc2_train, las=1, pch=16, col=rgb(0,0,1,.25), xlab="Jahresmitteltemperatur [°C/10]", ylab="Baumbedeckungsgrad [%]", xlim=c(-52, 400), main="Polynome 2. Grades", cex=.1+ sqrt( BIO12)/20)
lines(pred.temp.poly$fit ~ newD.temp$BIO1, col="red", lwd=2)
lines(pred.temp.poly$upr ~ newD.temp$BIO1, col="red", lty=2, lwd=2)
lines(pred.temp.poly$lwr ~ newD.temp$BIO1, col="red", lty=2, lwd=2)
lines(pred.temp.p600.poly$fit ~ newD.temp$BIO1, col="orange", lwd=2)
lines(pred.temp.p600.poly$upr ~ newD.temp$BIO1, col="orange", lty=2, lwd=2)
lines(pred.temp.p600.poly$lwr ~ newD.temp$BIO1, col="orange", lty=2, lwd=2)
lines(pred.temp.p1400.poly$fit ~ newD.temp$BIO1, col="firebrick4", lwd=2)
lines(pred.temp.p1400.poly$upr ~ newD.temp$BIO1, col="firebrick4", lty=2, lwd=2)
lines(pred.temp.p1400.poly$lwr ~ newD.temp$BIO1, col="firebrick4", lty=2, lwd=2)
legend(x="right", legend=c("600", "1057.1", "1400"), title = "Jahresniederschlag", col=c("orange", "red", "firebrick4"), lwd=2, bty='n')
legend(x=320, y=28, legend=paste(prcpVals, "mm"), pt.cex=.1+ sqrt(prcpVals)/20, pch=16, col="blue", bty='n')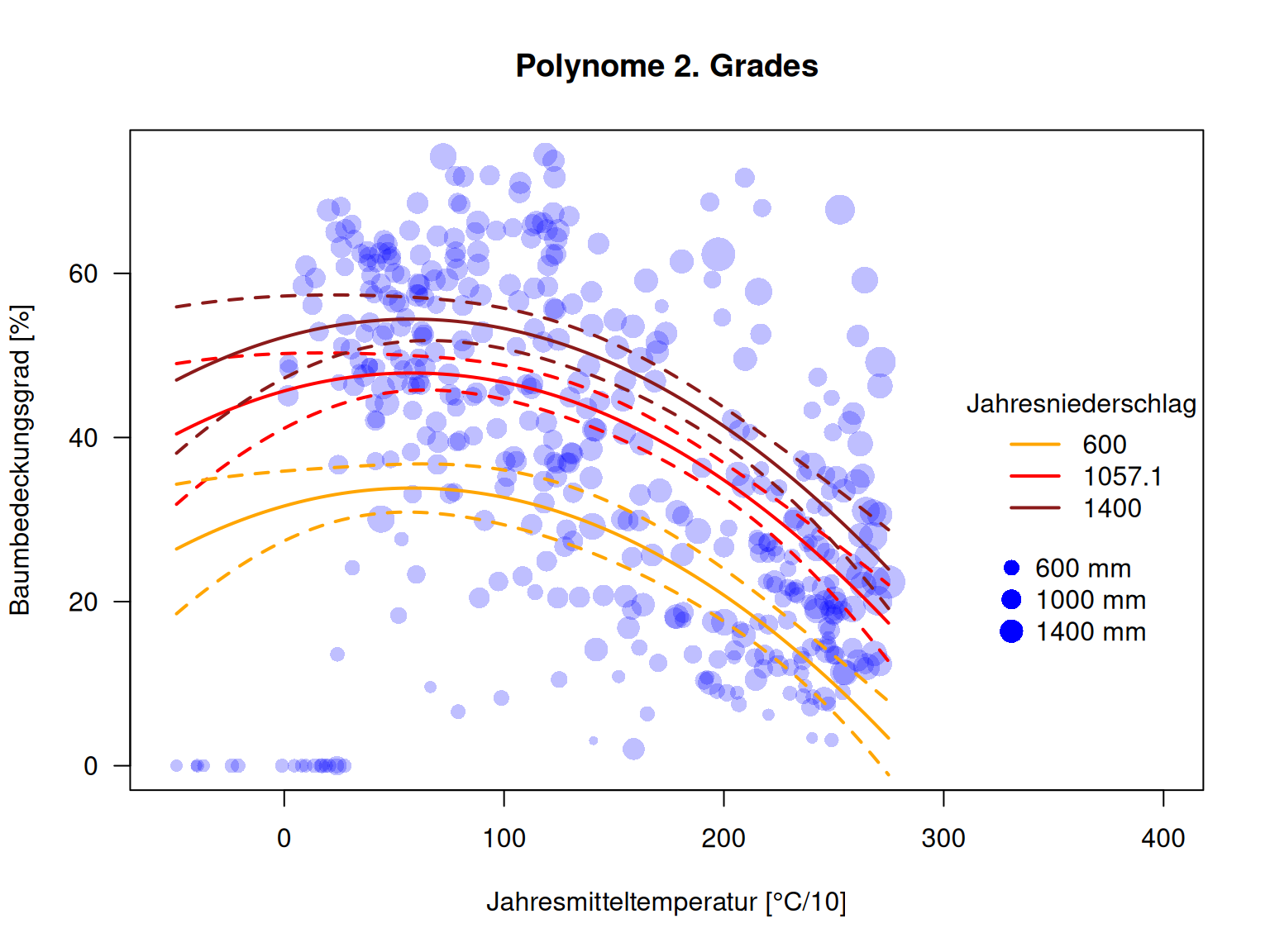
plot(MOD44B ~ BIO12, data=dat.glc2_train, las=1, pch=16, col=rgb(0,0,1,.25), xlab="Jahresniederschlag [mm]", ylab="Baumbedeckungsgrad [%]", xlim=c(0, 4000), main="Polynome 2. Grades", cex=.1+ sqrt(52+BIO1)/10)
lines(pred.prcp.poly$fit ~ newD.prcp$BIO12, col="red", lwd=2)
lines(pred.prcp.poly$upr ~ newD.prcp$BIO12, col="red", lty=2, lwd=2)
lines(pred.prcp.poly$lwr ~ newD.prcp$BIO12, col="red", lty=2, lwd=2)
lines(pred.prcp.t60.poly$fit ~ newD.prcp$BIO12, col="orange", lwd=2)
lines(pred.prcp.t60.poly$upr ~ newD.prcp$BIO12, col="orange", lty=2, lwd=2)
lines(pred.prcp.t60.poly$lwr ~ newD.prcp$BIO12, col="orange", lty=2, lwd=2)
lines(pred.prcp.t250.poly$fit ~ newD.prcp$BIO12, col="firebrick4", lwd=2)
lines(pred.prcp.t250.poly$upr ~ newD.prcp$BIO12, col="firebrick4", lty=2, lwd=2)
lines(pred.prcp.t250.poly$lwr ~ newD.prcp$BIO12, col="firebrick4", lty=2, lwd=2)
legend(x="right", legend=c("6", "14", "25"), title = "Jahresmittel-\ntemperatur [°C]", col=c("orange", "red", "firebrick4"), lwd=2, bty='n')
tempVals <- seq(-5, 30, by=10)
legend(x=3400, y=30, legend=paste(tempVals, "°C"), pt.cex=.1+ sqrt(52+tempVals*10)/10, pch=16, col="blue", bty='n')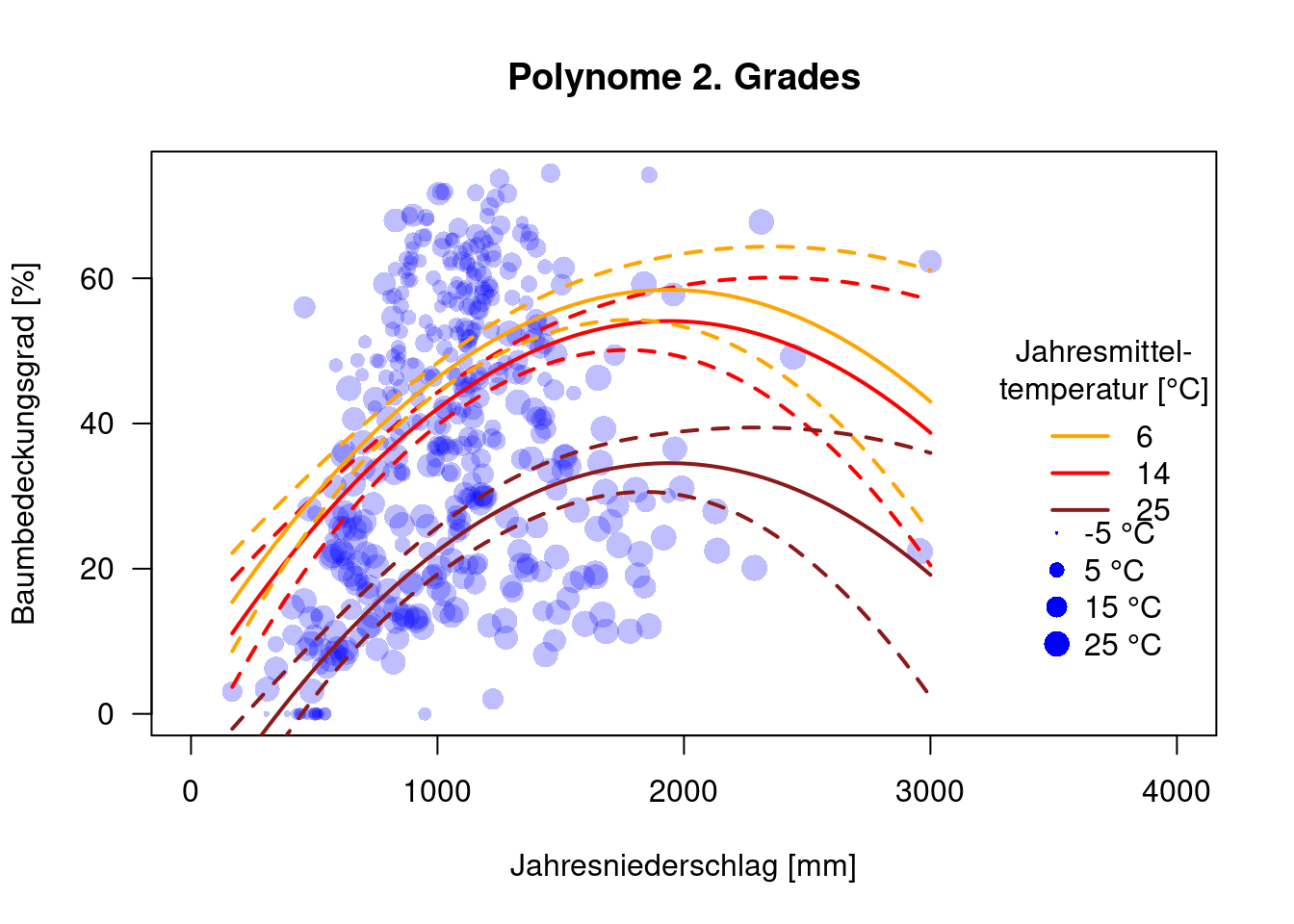
Wir sehen, dass bei hohen Niederschlagsmengen die Unsicherheit aufgrund der geringen Datendichte sehr hoch ist.
15.4.5 Polynomiale Terme und Interaktionen
##
## Call:
## lm(formula = MOD44B ~ poly(BIO1, 2) * poly(BIO12, 2), data = dat.glc2_train)
##
## Residuals:
## Min 1Q Median 3Q Max
## -55.199 -9.403 -0.710 8.980 43.793
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 36.1750 0.7982 45.320 < 2e-16 ***
## poly(BIO1, 2)1 -193.4915 17.7601 -10.895 < 2e-16 ***
## poly(BIO1, 2)2 -12.5650 17.0523 -0.737 0.46162
## poly(BIO12, 2)1 109.0577 24.2838 4.491 9.16e-06 ***
## poly(BIO12, 2)2 -174.4439 25.9143 -6.732 5.48e-11 ***
## poly(BIO1, 2)1:poly(BIO12, 2)1 12.1722 545.4537 0.022 0.98221
## poly(BIO1, 2)2:poly(BIO12, 2)1 -891.6913 391.6951 -2.276 0.02332 *
## poly(BIO1, 2)1:poly(BIO12, 2)2 3698.6353 544.0595 6.798 3.62e-11 ***
## poly(BIO1, 2)2:poly(BIO12, 2)2 -955.8178 275.6678 -3.467 0.00058 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 13.58 on 423 degrees of freedom
## Multiple R-squared: 0.5374, Adjusted R-squared: 0.5287
## F-statistic: 61.43 on 8 and 423 DF, p-value: < 2.2e-16## Single term deletions
##
## Model:
## MOD44B ~ poly(BIO1, 2) * poly(BIO12, 2)
## Df Sum of Sq RSS AIC
## <none> 78037 2299.5
## poly(BIO1, 2):poly(BIO12, 2) 4 21606 99643 2380.8Der BIC empfiehlt, die Interaktion im Modell zu lassen.
Vorhersage
pred.prcp.int.poly <- as.data.frame(predict(g.int.poly, newdata = newD.prcp, interval="confidence"))
pred.prcp.t60.int.poly <- as.data.frame(predict(g.int.poly, newdata = newD.prcp.t60, interval="confidence"))
pred.prcp.t250.int.poly <- as.data.frame(predict(g.int.poly, newdata = newD.prcp.t250, interval="confidence"))
pred.temp.int.poly <- as.data.frame(predict(g.int.poly, newdata = newD.temp, interval="confidence"))
pred.temp.p600.int.poly <- as.data.frame(predict(g.int.poly, newdata = newD.temp.p600, interval="confidence"))
pred.temp.p1400.int.poly <- as.data.frame(predict(g.int.poly, newdata = newD.temp.p1400, interval="confidence"))Plotten der Zusammenhänge
plot(MOD44B ~ BIO1, data=dat.glc2_train, las=1, pch=16, col=rgb(0,0,1,.25), xlab="Jahresmitteltemperatur [°C/10]", ylab="Baumbedeckungsgrad [%]", xlim=c(-52, 400), main="Interaktion und Polynome 2. Grades", cex=.1+ sqrt( BIO12)/20)
lines(pred.temp.int.poly$fit ~ newD.temp$BIO1, col="red", lwd=2)
lines(pred.temp.int.poly$upr ~ newD.temp$BIO1, col="red", lty=2, lwd=2)
lines(pred.temp.int.poly$lwr ~ newD.temp$BIO1, col="red", lty=2, lwd=2)
lines(pred.temp.p600.int.poly$fit ~ newD.temp$BIO1, col="orange", lwd=2)
lines(pred.temp.p600.int.poly$upr ~ newD.temp$BIO1, col="orange", lty=2, lwd=2)
lines(pred.temp.p600.int.poly$lwr ~ newD.temp$BIO1, col="orange", lty=2, lwd=2)
lines(pred.temp.p1400.int.poly$fit ~ newD.temp$BIO1, col="firebrick4", lwd=2)
lines(pred.temp.p1400.int.poly$upr ~ newD.temp$BIO1, col="firebrick4", lty=2, lwd=2)
lines(pred.temp.p1400.int.poly$lwr ~ newD.temp$BIO1, col="firebrick4", lty=2, lwd=2)
legend(x="right", legend=c("600", "1057.1", "1400"), title = "Jahresniederschlag", col=c("orange", "red", "firebrick4"), lwd=2, bty='n')
legend(x=320, y=28, legend=paste(prcpVals, "mm"), pt.cex=.1+ sqrt(prcpVals)/20, pch=16, col="blue", bty='n')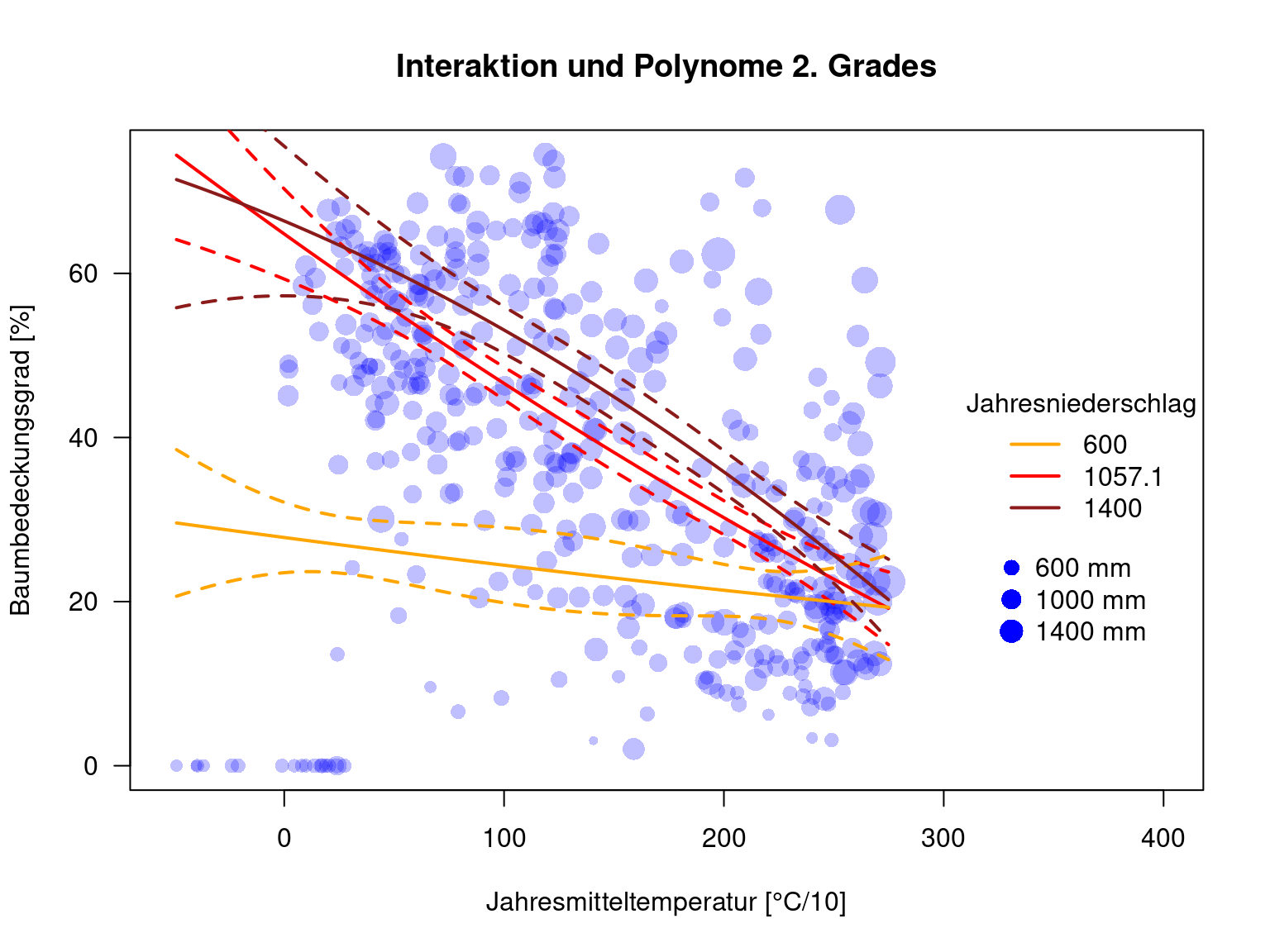
plot(MOD44B ~ BIO12, data=dat.glc2_train, las=1, pch=16, col=rgb(0,0,1,.25), xlab="Jahresniederschlag [mm]", ylab="Baumbedeckungsgrad [%]", xlim=c(0, 4000), main="Interaktion und Polynome 2. Grades", cex=.1+ sqrt(52+BIO1)/10)
lines(pred.prcp.int.poly$fit ~ newD.prcp$BIO12, col="red", lwd=2)
lines(pred.prcp.int.poly$upr ~ newD.prcp$BIO12, col="red", lty=2, lwd=2)
lines(pred.prcp.int.poly$lwr ~ newD.prcp$BIO12, col="red", lty=2, lwd=2)
lines(pred.prcp.t60.int.poly$fit ~ newD.prcp$BIO12, col="orange", lwd=2)
lines(pred.prcp.t60.int.poly$upr ~ newD.prcp$BIO12, col="orange", lty=2, lwd=2)
lines(pred.prcp.t60.int.poly$lwr ~ newD.prcp$BIO12, col="orange", lty=2, lwd=2)
lines(pred.prcp.t250.int.poly$fit ~ newD.prcp$BIO12, col="firebrick4", lwd=2)
lines(pred.prcp.t250.int.poly$upr ~ newD.prcp$BIO12, col="firebrick4", lty=2, lwd=2)
lines(pred.prcp.t250.int.poly$lwr ~ newD.prcp$BIO12, col="firebrick4", lty=2, lwd=2)
legend(x="right", legend=c("6", "14", "25"), title = "Jahresmittel-\ntemperatur [°C]", col=c("orange", "red", "firebrick4"), lwd=2, bty='n')
tempVals <- seq(-5, 30, by=10)
legend(x=3400, y=30, legend=paste(tempVals, "°C"), pt.cex=.1+ sqrt(52+tempVals*10)/10, pch=16, col="blue", bty='n')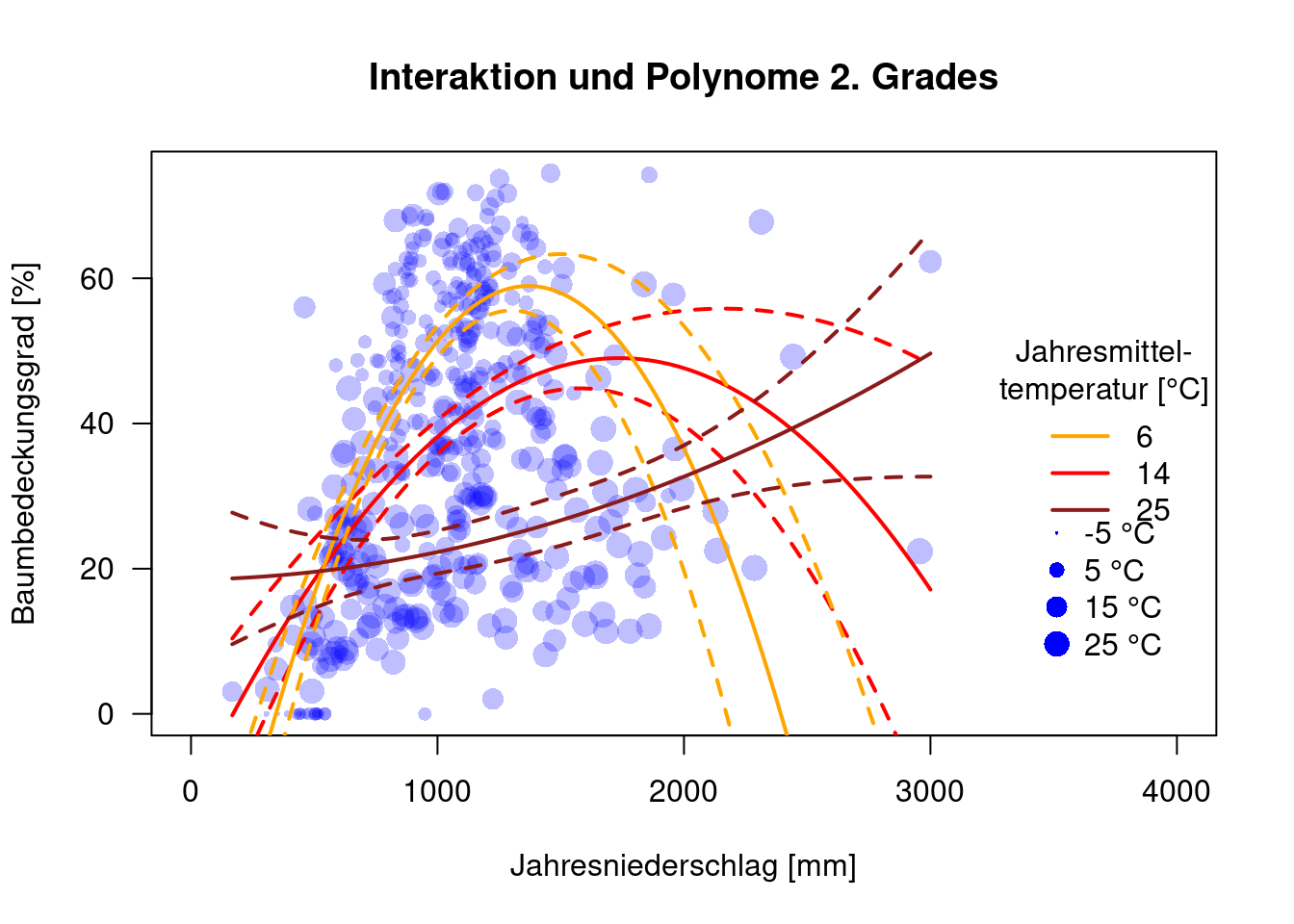
Für hohe Jahresniederschläge haben wir etwas wenig Daten, so dass mir die Kurvenverläufe für hohe Temperaturen nicht gut gefallen.
15.4.6 Polynomiale Terme und Interaktionen
##
## Call:
## lm(formula = MOD44B ~ BIO1 * poly(BIO12, 2), data = dat.glc2_train)
##
## Residuals:
## Min 1Q Median 3Q Max
## -52.978 -9.451 -1.155 9.330 43.319
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 5.396e+01 1.500e+00 35.982 < 2e-16 ***
## BIO1 -1.236e-01 8.866e-03 -13.946 < 2e-16 ***
## poly(BIO12, 2)1 2.485e+02 4.794e+01 5.183 3.38e-07 ***
## poly(BIO12, 2)2 -3.586e+02 4.614e+01 -7.770 5.91e-14 ***
## BIO1:poly(BIO12, 2)1 -7.593e-01 2.139e-01 -3.549 0.000429 ***
## BIO1:poly(BIO12, 2)2 1.505e+00 2.043e-01 7.368 9.08e-13 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 13.74 on 426 degrees of freedom
## Multiple R-squared: 0.5231, Adjusted R-squared: 0.5175
## F-statistic: 93.47 on 5 and 426 DF, p-value: < 2.2e-16## Single term deletions
##
## Model:
## MOD44B ~ BIO1 * poly(BIO12, 2)
## Df Sum of Sq RSS AIC
## <none> 80448 2294.4
## BIO1:poly(BIO12, 2) 2 24510 104958 2397.2Der BIC empfiehlt, die Interaktion im Modell zu lassen.
Vorhersage
pred.prcp.int.poly.prc <- as.data.frame(predict(g.int.polyPrc, newdata = newD.prcp, interval="confidence"))
pred.prcp.t60.int.poly.prc <- as.data.frame(predict(g.int.polyPrc, newdata = newD.prcp.t60, interval="confidence"))
pred.prcp.t250.int.poly.prc <- as.data.frame(predict(g.int.polyPrc, newdata = newD.prcp.t250, interval="confidence"))
pred.temp.int.poly.prc <- as.data.frame(predict(g.int.polyPrc, newdata = newD.temp, interval="confidence"))
pred.temp.p600.int.poly.prc <- as.data.frame(predict(g.int.polyPrc, newdata = newD.temp.p600, interval="confidence"))
pred.temp.p1400.int.poly.prc <- as.data.frame(predict(g.int.polyPrc, newdata = newD.temp.p1400, interval="confidence"))Plotten der Zusammenhänge
plot(MOD44B ~ BIO1, data=dat.glc2_train, las=1, pch=16, col=rgb(0,0,1,.25), xlab="Jahresmitteltemperatur [°C/10]", ylab="Baumbedeckungsgrad [%]", xlim=c(-52, 400), main="Interaktion und Polynome 2. Grades", cex=.1+ sqrt( BIO12)/20)
lines(pred.temp.int.poly.prc$fit ~ newD.temp$BIO1, col="red", lwd=2)
lines(pred.temp.int.poly.prc$upr ~ newD.temp$BIO1, col="red", lty=2, lwd=2)
lines(pred.temp.int.poly.prc$lwr ~ newD.temp$BIO1, col="red", lty=2, lwd=2)
lines(pred.temp.p600.int.poly.prc$fit ~ newD.temp$BIO1, col="orange", lwd=2)
lines(pred.temp.p600.int.poly.prc$upr ~ newD.temp$BIO1, col="orange", lty=2, lwd=2)
lines(pred.temp.p600.int.poly.prc$lwr ~ newD.temp$BIO1, col="orange", lty=2, lwd=2)
lines(pred.temp.p1400.int.poly.prc$fit ~ newD.temp$BIO1, col="firebrick4", lwd=2)
lines(pred.temp.p1400.int.poly.prc$upr ~ newD.temp$BIO1, col="firebrick4", lty=2, lwd=2)
lines(pred.temp.p1400.int.poly.prc$lwr ~ newD.temp$BIO1, col="firebrick4", lty=2, lwd=2)
legend(x="right", legend=c("600", "1057.1", "1400"), title = "Jahresniederschlag", col=c("orange", "red", "firebrick4"), lwd=2, bty='n')
legend(x=320, y=28, legend=paste(prcpVals, "mm"), pt.cex=.1+ sqrt(prcpVals)/20, pch=16, col="blue", bty='n')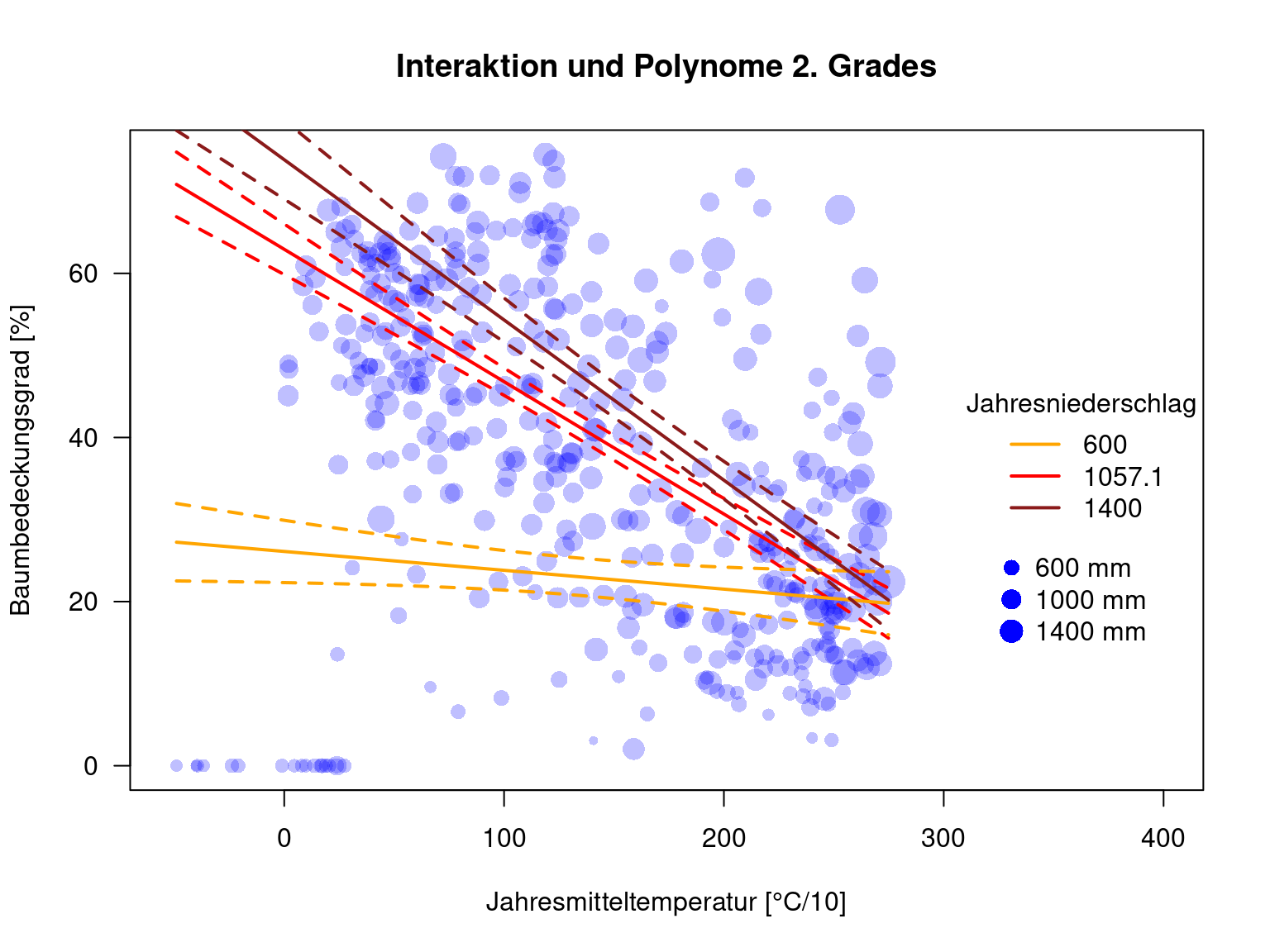
plot(MOD44B ~ BIO12, data=dat.glc2_train, las=1, pch=16, col=rgb(0,0,1,.25), xlab="Jahresniederschlag [mm]", ylab="Baumbedeckungsgrad [%]", xlim=c(0, 4000), main="Interaktion und Polynome 2. Grades", cex=.1+ sqrt(52+BIO1)/10)
lines(pred.prcp.int.poly.prc$fit ~ newD.prcp$BIO12, col="red", lwd=2)
lines(pred.prcp.int.poly.prc$upr ~ newD.prcp$BIO12, col="red", lty=2, lwd=2)
lines(pred.prcp.int.poly.prc$lwr ~ newD.prcp$BIO12, col="red", lty=2, lwd=2)
lines(pred.prcp.t60.int.poly.prc$fit ~ newD.prcp$BIO12, col="orange", lwd=2)
lines(pred.prcp.t60.int.poly.prc$upr ~ newD.prcp$BIO12, col="orange", lty=2, lwd=2)
lines(pred.prcp.t60.int.poly.prc$lwr ~ newD.prcp$BIO12, col="orange", lty=2, lwd=2)
lines(pred.prcp.t250.int.poly.prc$fit ~ newD.prcp$BIO12, col="firebrick4", lwd=2)
lines(pred.prcp.t250.int.poly.prc$upr ~ newD.prcp$BIO12, col="firebrick4", lty=2, lwd=2)
lines(pred.prcp.t250.int.poly.prc$lwr ~ newD.prcp$BIO12, col="firebrick4", lty=2, lwd=2)
legend(x="right", legend=c("6", "14", "25"), title = "Jahresmittel-\ntemperatur [°C]", col=c("orange", "red", "firebrick4"), lwd=2, bty='n')
tempVals <- seq(-5, 30, by=10)
legend(x=3400, y=28, legend=paste(tempVals, "°C"), pt.cex=.1+ sqrt(52+tempVals*10)/10, pch=16, col="blue", bty='n')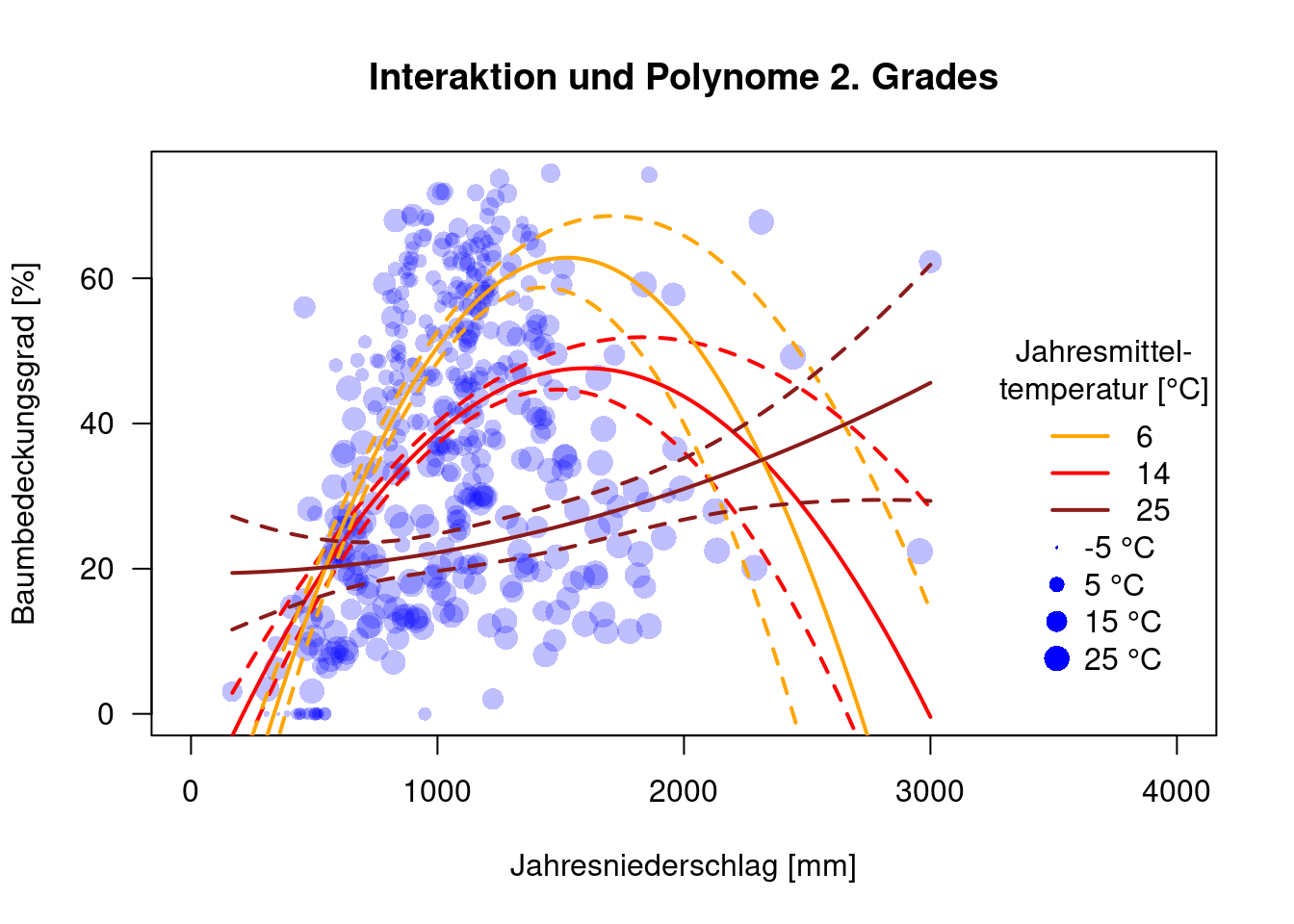
Mann könnte auch überlegen, die Interaktion nur zwischen den beiden Haupteffekten zuzulassen.
15.4.7 Generalisierungsfehler
Nun wollen wir aufgrundlage des Testdatensatzes den Generalisierungsfehler betrachten und schauen, ob die Modelle auch mit unbekannten Daten klarkommt. In einer realen Anwendung müsste man sich zudem um die räunmliche Autokorrelation kümmern, was die Signifikanzniveaus aber auch den Generalisierungs negativ verändern dürfte.
test.main <- predict(g.main, newdata = dat.glc2_test)
test.poly <- predict(g.poly, newdata = dat.glc2_test)
test.int <- predict(g.int, newdata = dat.glc2_test)
test.int.poly <- predict(g.int.poly, newdata = dat.glc2_test)
test.int.polyPrc <- predict(g.int.polyPrc, newdata = dat.glc2_test)
train.main <- predict(g.main, newdata = dat.glc2_train)
train.poly <- predict(g.poly, newdata = dat.glc2_train)
train.int <- predict(g.int, newdata = dat.glc2_train)
train.int.poly <- predict(g.int.poly, newdata = dat.glc2_train)
train.int.polyPrc <- predict(g.int.polyPrc, newdata = dat.glc2_train)Funktion zum Berechnen des root mean square errors (der Quadratwurzel des mittleren quadrierten Fehlers):
res <- data.frame(model=c("Haupteffekte", "Interaktion", "Polynom 2. Grades", "Quadratisch &\nInteraktion", "Niederschlag quadartisch,\n Temperatur und Interaktion"), rsmeTrain = NA, rsmeTest=NA)
res$rsmeTest[1] <- rsme(dat.glc2_test$MOD44B, test.main)
res$rsmeTest[2] <- rsme(dat.glc2_test$MOD44B, test.int)
res$rsmeTest[3] <- rsme(dat.glc2_test$MOD44B, test.poly)
res$rsmeTest[4] <- rsme(dat.glc2_test$MOD44B, test.int.poly)
res$rsmeTest[5] <- rsme(dat.glc2_test$MOD44B, test.int.polyPrc)
res$rsmeTrain[1] <- rsme(dat.glc2_train$MOD44B, train.main)
res$rsmeTrain[2] <- rsme(dat.glc2_train$MOD44B, train.int)
res$rsmeTrain[3] <- rsme(dat.glc2_train$MOD44B, train.poly)
res$rsmeTrain[4] <- rsme(dat.glc2_train$MOD44B, train.int.poly)
res$rsmeTrain[5] <- rsme(dat.glc2_train$MOD44B, train.int.polyPrc)Je kleiner der RSME, desto besser.
ggplot(data= res, mapping = aes(x= model, y= rsmeTest)) +
geom_point() +
coord_flip() + ylab("RSME") + xlab("") +
labs(title = "Validierungsdatensatz")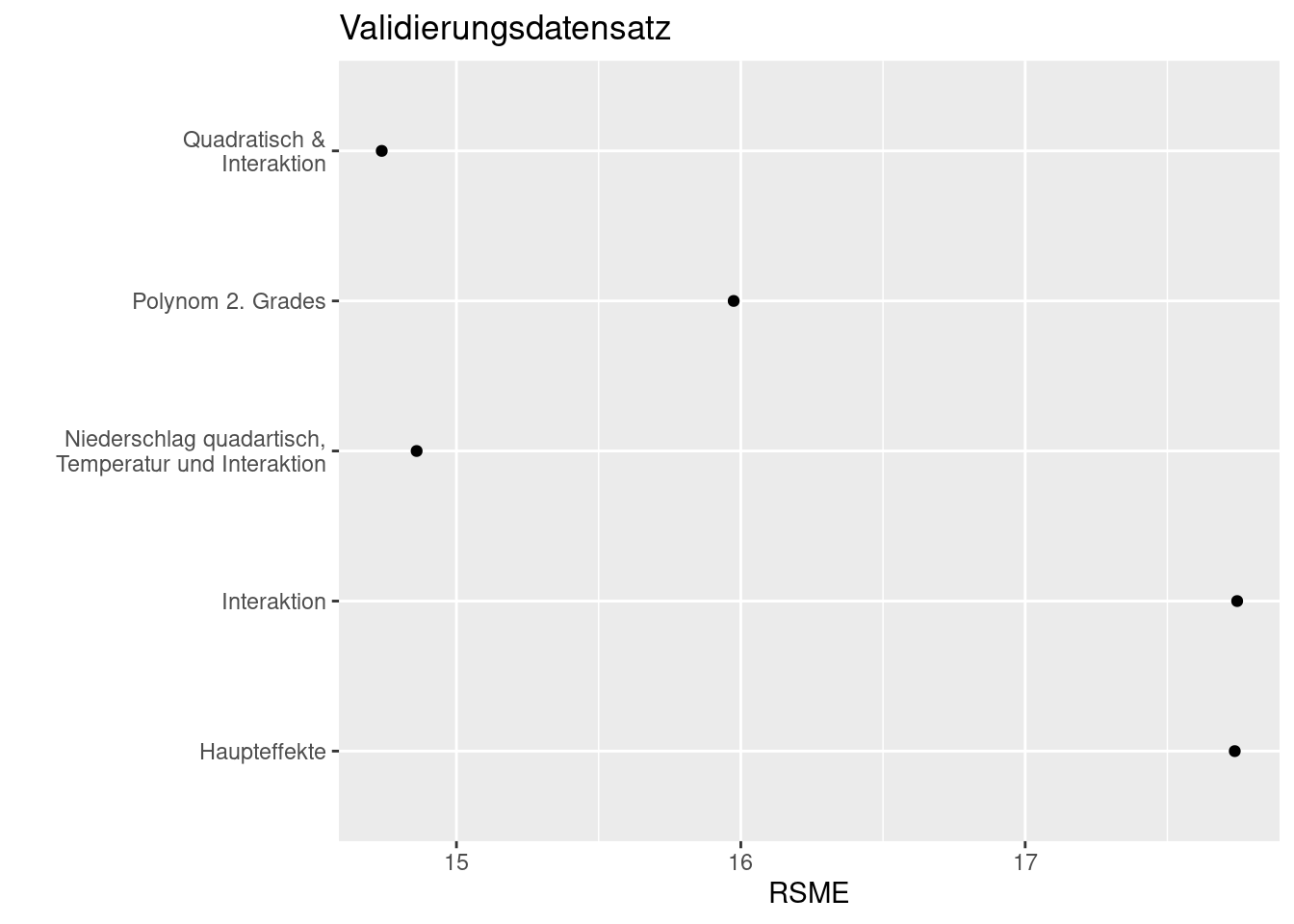
Beim Vergleich mit den Testdaten erkennen wir, dass das Modell mit quadratischem Term bei Niederschlag, dem Haupteffekt für Temperatur und Interaktionen am besten abschneide. Wir sehen hier auch, dass dieses Modell mit der Interaktion und dem quadratischem Effekt deutlich besser generalisiert als das quadratische Modell ohne Interaktionen- beim Trainingsdatensatz sind beide Modelle gleich gut. Der Fehler von 13.5% Waldbedeckung ist allerdings erheblich.
ggplot(data= res, mapping = aes(x= model, y= rsmeTrain)) +
geom_point() +
coord_flip() + ylab("RSME") + xlab("") +
labs(title = "Trainingsdatensatz")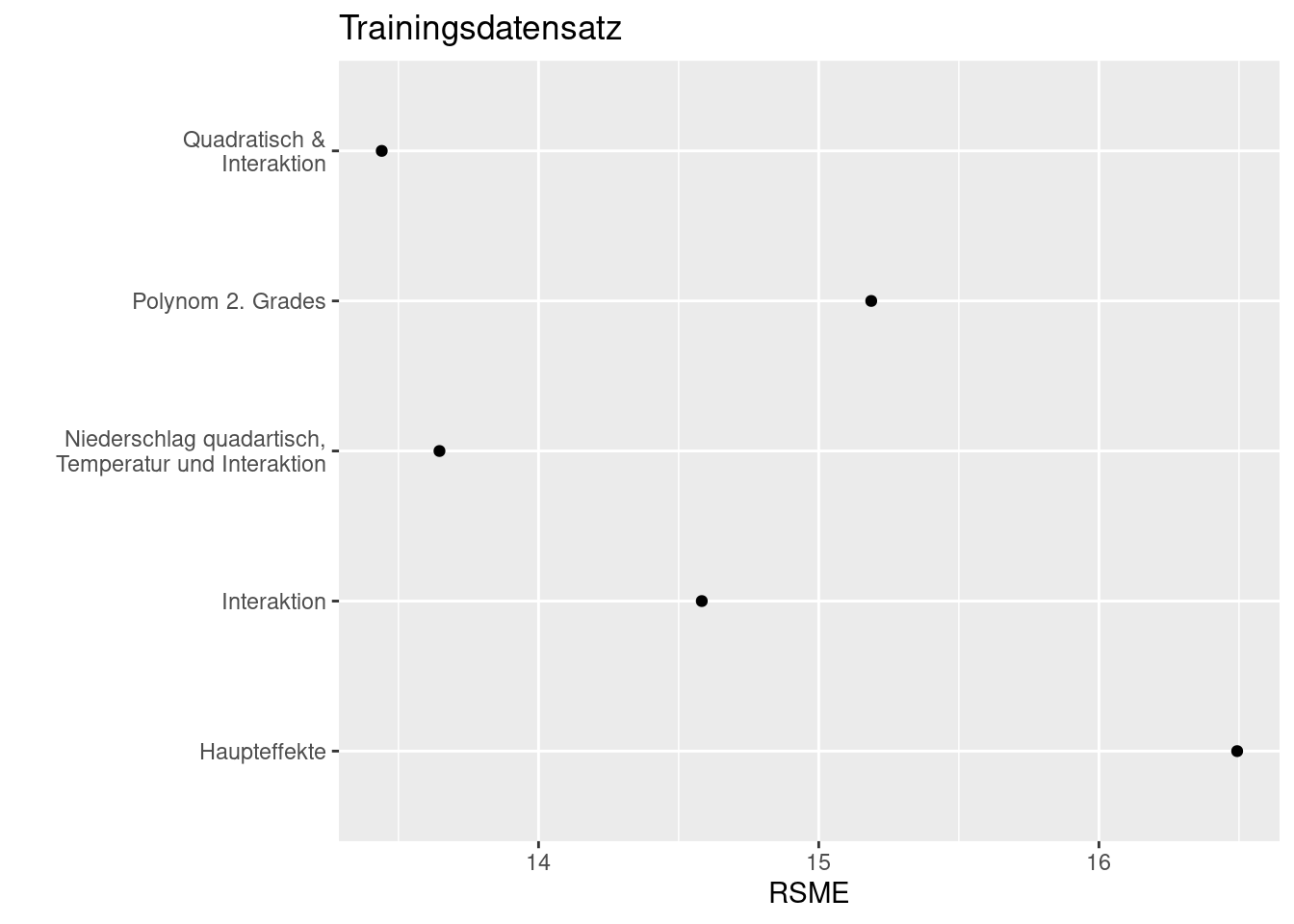
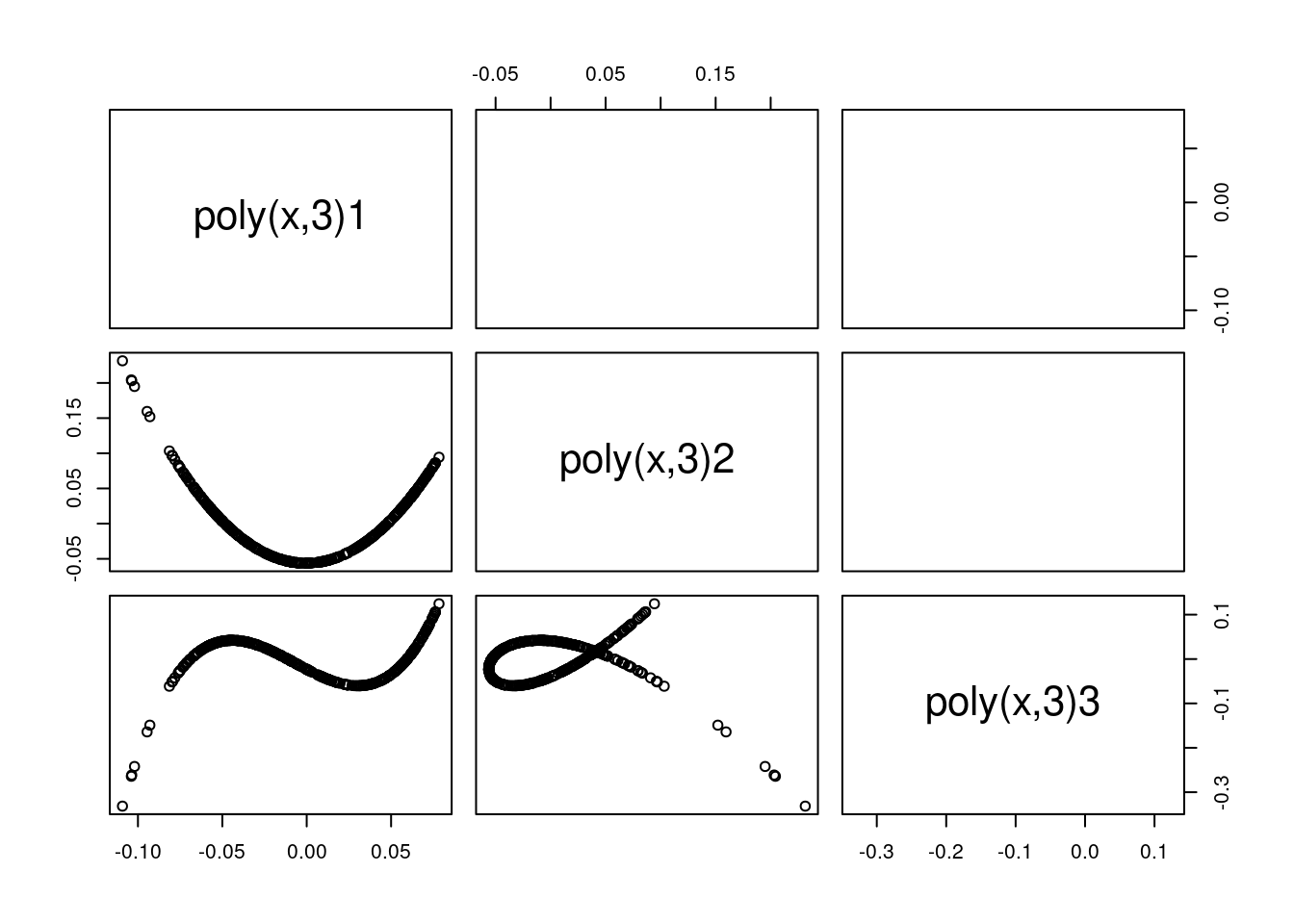
Poly erzeugt voneinander unabhängige (orthogonale) Variablen - die Korrelationskoeffizienten sind quasi 0.
Im Gegensatz dazu sind x, \(x^2\) und \(x^3\) hochgradig korreliert.
pairs(cbind(dat.glc2_train$BIO1, dat.glc2_train$BIO1^2, dat.glc2_train$BIO1^3 ), upper.panel = panel.cor, labels=c( "x", "x^2", "x^3"))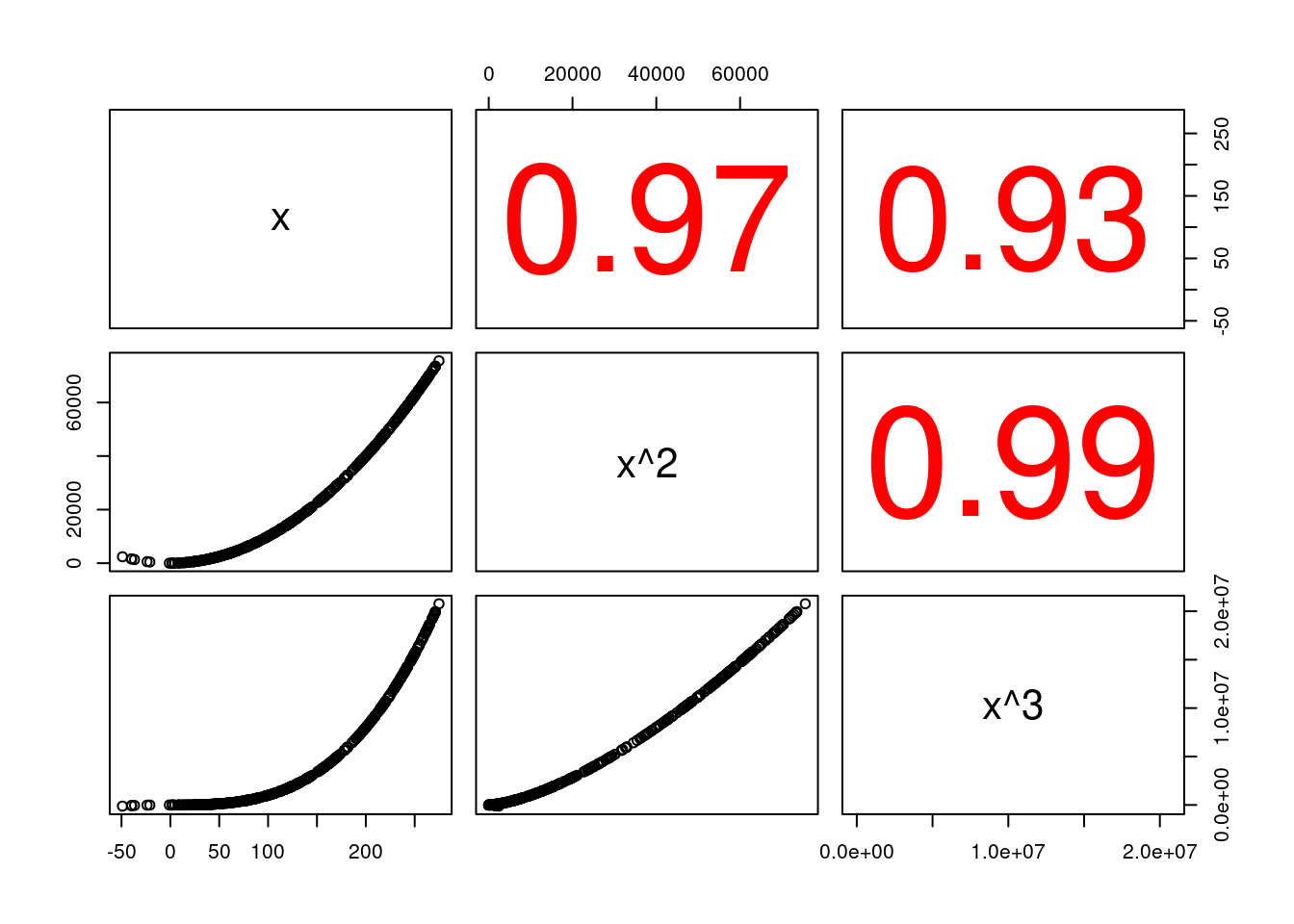
Das erste orthogonale Polygon ist dabei (muss!) perfekt mit x korreliert.
pairs(cbind(bio1.poly3, dat.glc2_train$BIO1, dat.glc2_train$BIO1^2, dat.glc2_train$BIO1^3 ), upper.panel = panel.cor, labels=c("poly(x,3)1", "poly(x,3)2", "poly(x,3)3", "x", "x^2", "x^3"))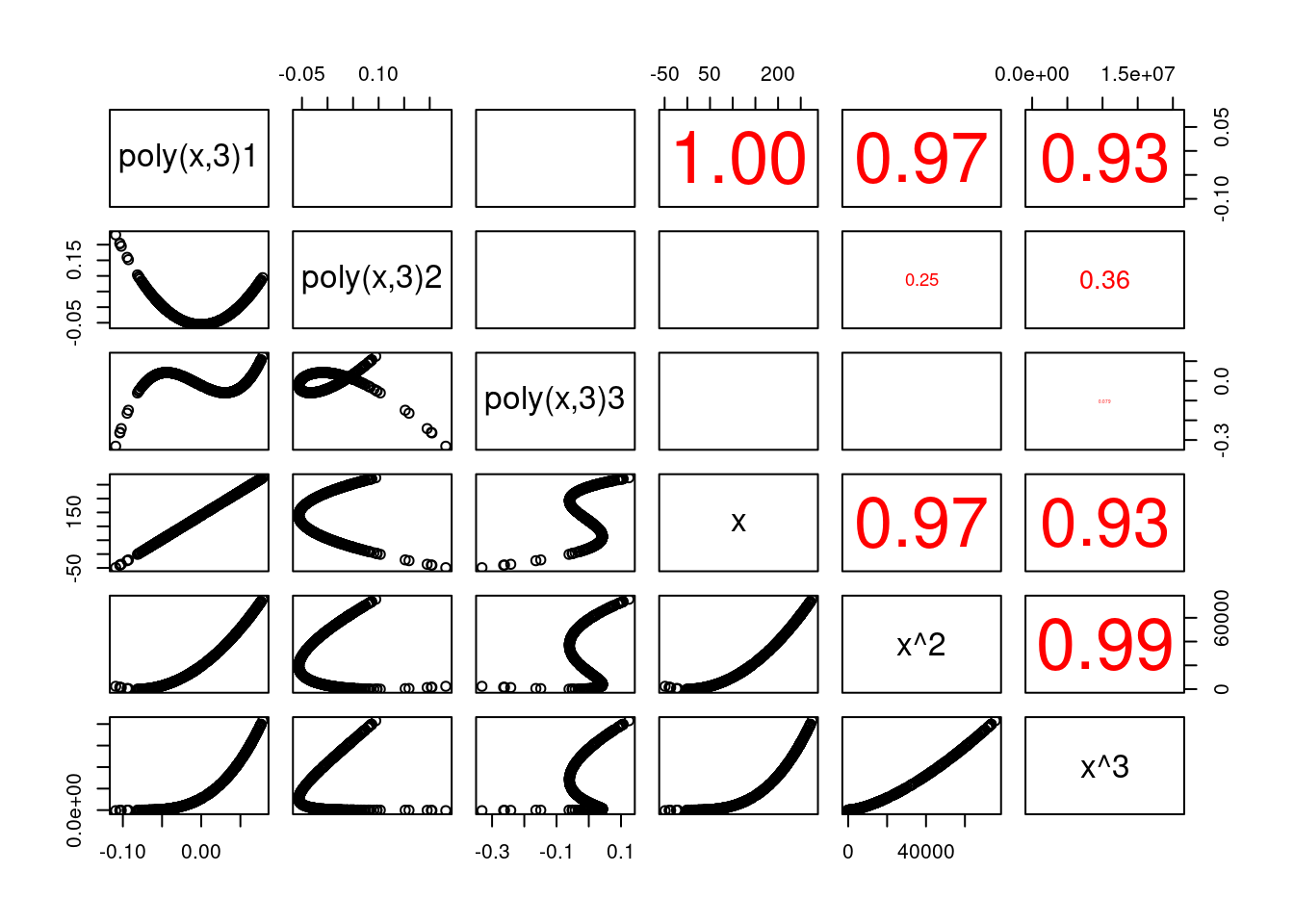
Erzeugen kann man die othronomalen Polygone als QR Zerlegung einer Matrix, die in jeder Spalte \(x^{spaltennummer}\) enthält, die anschließend zentriert wird.
# Matrix mit x, x^2, x^3
polyMat <- cbind(dat.glc2_train$BIO1, dat.glc2_train$BIO1^2, dat.glc2_train$BIO1^3)
# zentrieren
polyMat[,1] <- polyMat[,1] - mean(polyMat[,1])
polyMat[,2] <- polyMat[,2] - mean(polyMat[,2])
polyMat[,3] <- polyMat[,3] - mean(polyMat[,3])
# QR Zerlegung der Matiry
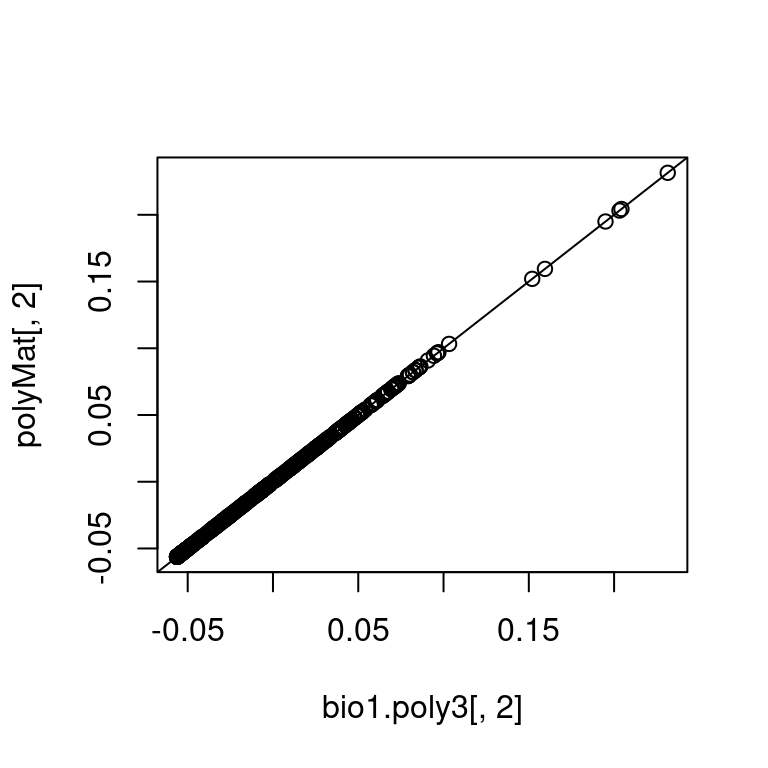
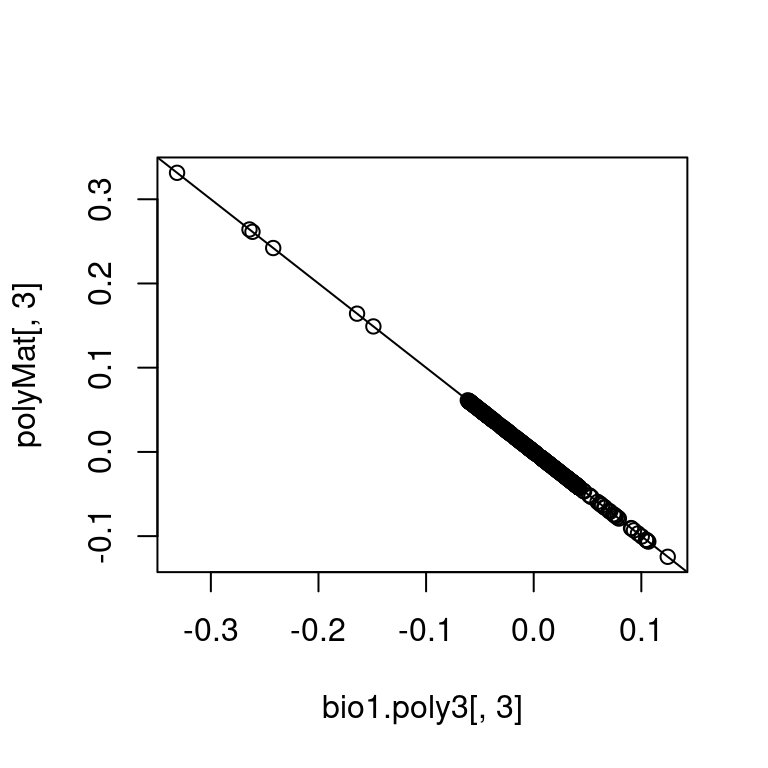
polyMat <- qr.Q(qr(polyMat))Die so gewonnenen Ergebnisse sind bis auf Rundungsfehler exakt dieselben wie die von poly berechneten.
## 1 2 3
## [1,] 1 0 0
## [2,] 0 1 0
## [3,] 0 0 -1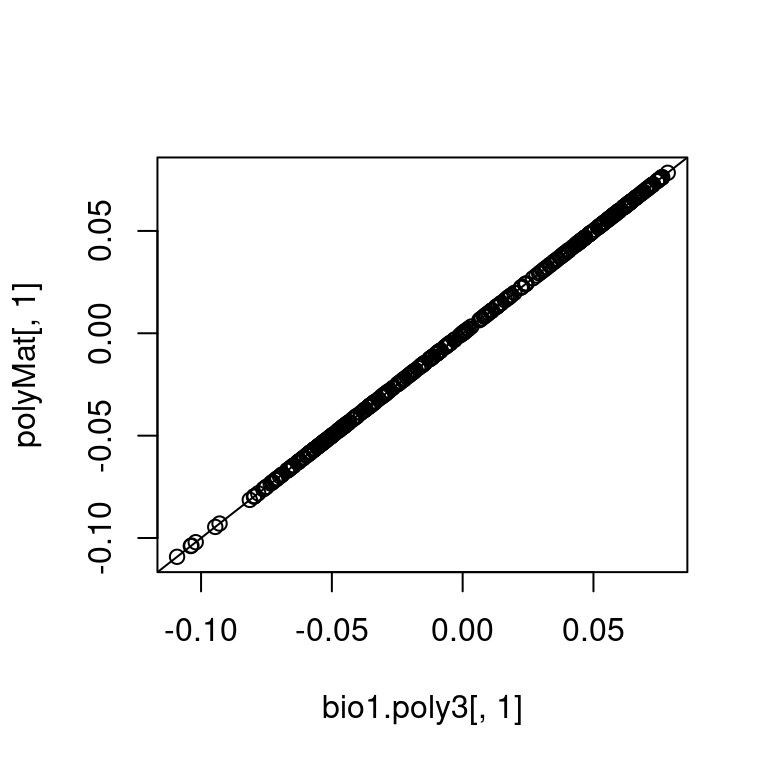
Weiterführende/zitierte Literatur
Eine Gerade ist ein Polynom ersten Grades.↩︎
In einem bestimmten Bereich, zu heiß ist auch nichts, quadratischer Effekt.↩︎
Auch hier wieder abhängig vom Pflanzentyp und innerhalb einer ökologischen Nische, die z.B. für Wasserlinien anders aussieht als für Kakteen.↩︎
Die original Daten sind unter folgendem Link abrufbar: [ftp://aftp.cmdl.noaa.gov/data/greenhouse_gases/co2/in-situ/surface/] ↩︎